Análisis residuales ajuste MA al precio interno del Café#
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import matplotlib.dates as mdates
from statsmodels.tsa.stattools import adfuller
from statsmodels.graphics.tsaplots import plot_acf, plot_pacf
from statsmodels.tsa.statespace.sarimax import SARIMAX
def plot_serie_tiempo(
serie: pd.DataFrame,
nombre: str,
unidades: str = None,
columna: str = None,
fecha_inicio: str = None,
fecha_fin: str = None,
color: str = 'navy',
linewidth: float = 2,
num_xticks: int = 12,
estacionalidad: str = None, # 'diciembre', 'enero', 'semana', 'semestre', 'custom_month'
custom_month: int = None, # Si quieres marcar otro mes (ejemplo: 3 para marzo)
vline_label: str = None, # Etiqueta para la(s) línea(s) vertical(es)
hlines: list = None, # lista de valores horizontales a marcar
hlines_labels: list = None, # lista de etiquetas para líneas horizontales
color_estacion: str = 'darkgray', # color de las líneas estacionales
alpha_estacion: float = 0.3, # transparencia de líneas estacionales
color_hline: str = 'gray', # color de las líneas horizontales
alpha_hline: float = 0.7 # transparencia de líneas horizontales
):
"""
Gráfico elegante de serie de tiempo.
- Eje X alineado con la primera fecha real de la serie.
- Opcional: marcar estacionalidades (diciembres, semanas, semestres, mes personalizado) con etiqueta.
- Líneas horizontales con etiqueta opcional (legend).
"""
df = serie.copy()
if columna is None:
columna = df.columns[0]
if fecha_inicio:
df = df[df.index >= fecha_inicio]
if fecha_fin:
df = df[df.index <= fecha_fin]
# Asegura que el índice sea datetime y esté ordenado
df = df.sort_index()
df.index = pd.to_datetime(df.index)
plt.style.use('ggplot')
fig, ax = plt.subplots(figsize=(14, 6))
# Gráfica principal
ax.plot(df.index, df[columna], color=color, linewidth=linewidth, label=nombre)
ax.set_title(f"Serie de tiempo: {nombre}", fontsize=20, weight='bold',
color='black')
ax.set_xlabel("Fecha", fontsize=15, color='black')
ax.set_ylabel(unidades, fontsize=15, color='black')
ax.tick_params(axis='both', colors='black', labelsize=13)
for label in ax.get_xticklabels() + ax.get_yticklabels():
label.set_color('black')
# Limita el rango del eje X exactamente al rango de fechas de la serie (no corrido)
ax.set_xlim(df.index.min(), df.index.max())
# Ticks equidistantes en eje X, asegurando que empieza en la primera fecha
idx = df.index
if len(idx) > num_xticks:
ticks = np.linspace(0, len(idx)-1, num_xticks, dtype=int)
ticks[0] = 0 # asegúrate que arranque en la primera fecha
ticklabels = [idx[i] for i in ticks]
ax.set_xticks(ticklabels)
ax.set_xticklabels([pd.to_datetime(t).strftime('%b %Y') for t in ticklabels], rotation=0, color='black')
else:
ax.xaxis.set_major_formatter(mdates.DateFormatter('%b %Y'))
fig.autofmt_xdate(rotation=0)
# ==============================
# LÍNEAS VERTICALES: Estacionalidad (con etiqueta en leyenda si se desea)
# ==============================
vlines_plotted = False
if estacionalidad is not None:
if estacionalidad == 'diciembre':
fechas_mark = df[df.index.month == 12].index
elif estacionalidad == 'enero':
fechas_mark = df[df.index.month == 1].index
elif estacionalidad == 'semana':
fechas_mark = df[df.index.weekday == 0].index
elif estacionalidad == 'semestre':
fechas_mark = df[df.index.month.isin([6, 12])].index
elif estacionalidad == 'custom_month' and custom_month is not None:
fechas_mark = df[df.index.month == custom_month].index
else:
fechas_mark = []
for i, f in enumerate(fechas_mark):
# Solo pone la etiqueta una vez (la primera línea)
if not vlines_plotted and vline_label is not None:
ax.axvline(f, color=color_estacion, alpha=alpha_estacion, linewidth=2, linestyle='--', zorder=0, label=vline_label)
vlines_plotted = True
else:
ax.axvline(f, color=color_estacion, alpha=alpha_estacion, linewidth=2, linestyle='--', zorder=0)
# ==============================
# LÍNEAS HORIZONTALES OPCIONALES, con leyenda
# ==============================
if hlines is not None:
if hlines_labels is None:
hlines_labels = [None] * len(hlines)
for i, h in enumerate(hlines):
if hlines_labels[i] is not None:
ax.axhline(h, color=color_hline, alpha=alpha_hline, linewidth=1.5, linestyle='--', zorder=0, label=hlines_labels[i])
else:
ax.axhline(h, color=color_hline, alpha=alpha_hline, linewidth=1.5, linestyle='--', zorder=0)
# Coloca la leyenda solo si hay etiquetas
handles, labels = ax.get_legend_handles_labels()
if any(labels):
ax.legend(loc='best', fontsize=13, frameon=True)
ax.grid(True, alpha=0.4)
plt.tight_layout()
plt.show()
##################################################################################
def analisis_estacionariedad(
serie: pd.Series,
nombre: str = None,
lags: int = 24,
xtick_interval: int = 3
):
"""
Gráfica y análisis de estacionariedad para una serie de tiempo:
- Serie original, diferencia, logaritmo y diferencia del logaritmo.
- Muestra la ACF, PACF y resultado ADF en subplots.
Args:
serie: Serie de tiempo (índice datetime, pandas.Series)
nombre: Nombre de la serie (para títulos)
lags: Número de rezagos para ACF/PACF
xtick_interval: Mostrar ticks en X cada este número de lags, incluyendo siempre el lag 1
"""
if nombre is None:
nombre = serie.name if serie.name is not None else "Serie"
# Transformaciones
serie_1 = serie.copy()
serie_2 = serie_1.diff().dropna()
serie_3 = np.log(serie_1)
serie_4 = serie_3.diff().dropna()
titulos = [
f"Serie original: {nombre}",
"Diferenciación",
"Logaritmo",
"Diferenciación del Logaritmo"
]
series = [serie_1, serie_2, serie_3, serie_4]
resultados_adf = []
interpretaciones = []
for i, serie_i in enumerate(series):
serie_ = serie_i.dropna()
# Selección de regresión en ADF
if i in [0, 2]:
adf = adfuller(serie_, regression='ct')
else:
adf = adfuller(serie_, regression='c')
estadistico = adf[0]
pvalue = adf[1]
resultados_adf.append((estadistico, pvalue))
interpretaciones.append("Estacionaria" if pvalue < 0.05 else "No estacionaria")
fig, axes = plt.subplots(4, 3, figsize=(18, 16))
colores = ['black', 'black', 'black', 'black']
for fila in range(4):
# Serie y etiquetas
axes[fila, 0].plot(series[fila], color=colores[fila])
axes[fila, 0].set_title(titulos[fila], color='black')
axes[fila, 0].set_xlabel("Fecha", color='black')
if fila == 0:
axes[fila, 0].set_ylabel("Valor", color='black')
elif fila == 1:
axes[fila, 0].set_ylabel("Δ Valor", color='black')
elif fila == 2:
axes[fila, 0].set_ylabel("Log(Valor)", color='black')
else:
axes[fila, 0].set_ylabel("Δ Log(Valor)", color='black')
axes[fila, 0].grid(True, alpha=0.3)
axes[fila, 0].tick_params(axis='both', labelsize=11, colors='black')
# ACF
plot_acf(
series[fila].dropna(),
lags=lags,
ax=axes[fila, 1],
zero=False,
color=colores[fila]
)
axes[fila, 1].set_title("ACF", color='black')
# xticks: incluir lag 1 y luego cada xtick_interval (ej: 1, 3, 6, ...)
xticks = [1] + list(range(xtick_interval, lags + 1, xtick_interval))
xticks = sorted(set(xticks)) # asegura que no haya duplicados
axes[fila, 1].set_xticks(xticks)
axes[fila, 1].tick_params(axis='both', labelsize=11, colors='black')
axes[fila, 1].set_xlabel("Lag", color='black')
axes[fila, 1].set_ylabel("Autocorrelación", color='black')
# PACF
plot_pacf(
series[fila].dropna(),
lags=lags,
ax=axes[fila, 2],
zero=False,
color=colores[fila]
)
axes[fila, 2].set_title("PACF", color='black')
axes[fila, 2].set_xticks(xticks)
axes[fila, 2].tick_params(axis='both', labelsize=11, colors='black')
axes[fila, 2].set_xlabel("Lag", color='black')
axes[fila, 2].set_ylabel("Autocorrelación parcial", color='black')
# Indicador estacionariedad (más abajo)
axes[fila, 0].text(
0.02, 0.85,
f"ADF: {resultados_adf[fila][0]:.2f}\np-valor: {resultados_adf[fila][1]:.4f}\n{interpretaciones[fila]}",
transform=axes[fila, 0].transAxes,
fontsize=11, bbox=dict(facecolor='white', alpha=0.85), color='black'
)
plt.tight_layout()
plt.show()
# Devuelve los resultados en un dict (opcional)
adf_dict = {
titulos[i]: {
"estadístico ADF": resultados_adf[i][0],
"p-valor": resultados_adf[i][1],
"interpretación": interpretaciones[i]
}
for i in range(4)
}
return adf_dict
Precio interno del Café#
# Cargar el archivo
serie = pd.read_excel("Precio_interno_cafe.xlsx")
# Corregir nombres de columnas si tienen espacios
serie.columns = serie.columns.str.strip()
# Convertir 'Fecha' a datetime y usar como índice
serie['Fecha'] = pd.to_datetime(serie['Fecha'])
serie.set_index('Fecha', inplace=True)
# Ordenar por fecha por si acaso
serie = serie.sort_index()
# Establecer frecuencia explícita para evitar el warning de statsmodels
serie.index.freq = serie.index.inferred_freq
serie.head()
| Precio | |
|---|---|
| Fecha | |
| 2000-01-01 | 371375.0 |
| 2000-02-01 | 354297.0 |
| 2000-03-01 | 360016.0 |
| 2000-04-01 | 347538.0 |
| 2000-05-01 | 353750.0 |
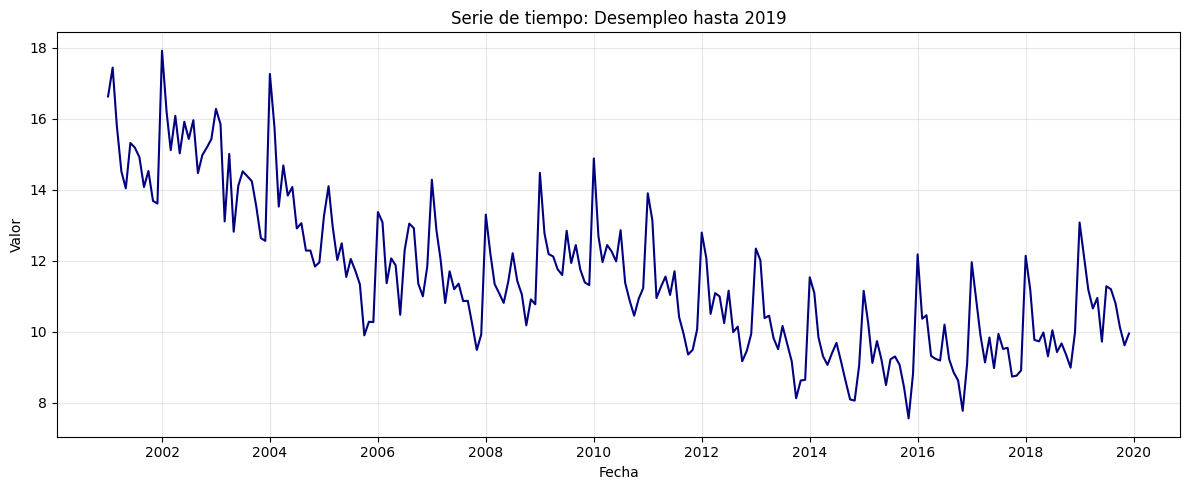
plot_serie_tiempo(
serie,
nombre="Precio interno del Café",
columna='Precio',
unidades='COP/carga',
estacionalidad='diciembre',
vline_label="Diciembre",
num_xticks = 14
)
adf_resultados = analisis_estacionariedad(
serie['Precio'],
nombre="Precio interno del Café",
lags=36,
xtick_interval=3
)
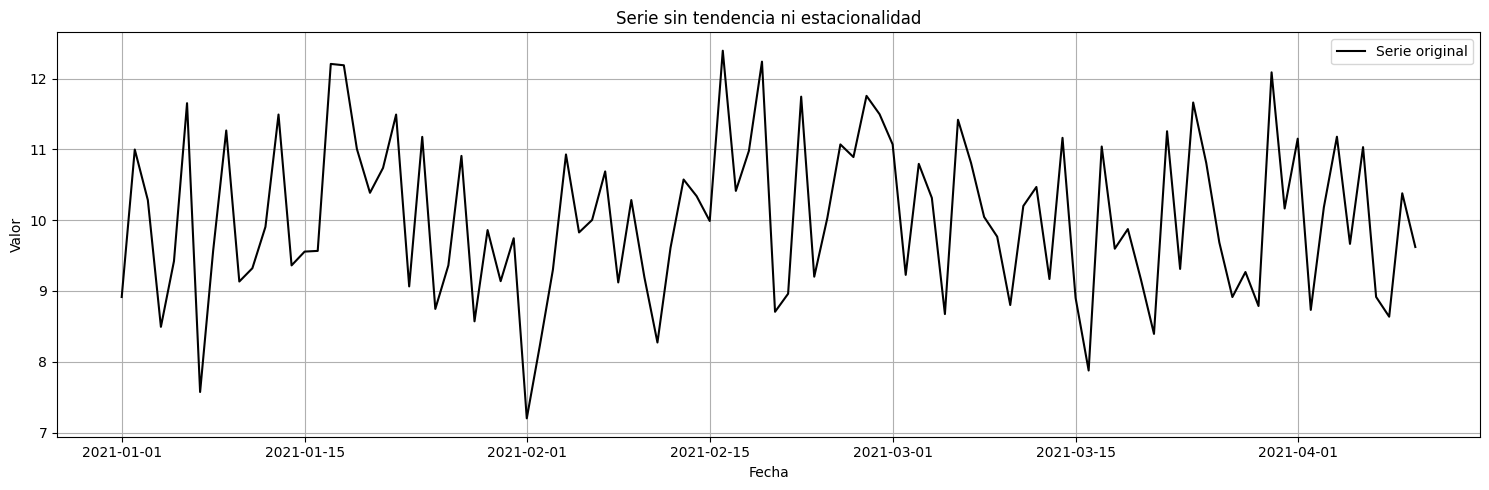
Modelo MA a la serie transformada: diff del logaritmo#
# Transformación: Logaritmo
df_log = np.log(serie)
plot_serie_tiempo(
df_log.diff().dropna(),
nombre="Primera diferencia de logaritmo del precio interno del Café",
columna='Precio',
unidades='',
num_xticks = 14
)
Se trabajará con la serie en logaritmo porque en el ajuste se indicará que se haga la primera diferencia
Conjunto de train y test:#
# Dividir en train y test (por ejemplo, 80% train, 20% test)
split = int(len(df_log) * 0.8)
train, test = df_log[:split], df_log[split:]
# Graficar train y test:
plt.figure(figsize=(12, 5))
plt.plot(train.diff().dropna(), label='Train', color='navy')
plt.plot(test.diff().dropna(), label='Test', color='orange')
plt.title("Conjunto de train y test")
plt.xlabel("Fecha")
plt.ylabel("Valor")
plt.legend()
plt.grid(True, alpha=0.3)
plt.tight_layout()
plt.show()
Ajuste MA(1)#
# Definir los parámetros del modelo AR (0, d, q)
order = (0, 1, 1) # Puedes ajustar según el análisis de ACF y PACF
trend = 'n' # 'c' = constante, 't' = tendencia, 'ct' = constante + tendencia, 'n' = sin tendencia
# Ajustar el modelo con los datos de entrenamiento
model = SARIMAX(train, order=order, trend=trend)
results = model.fit()
# Mostrar resumen del modelo
print(results.summary())
SARIMAX Results
==============================================================================
Dep. Variable: Precio No. Observations: 246
Model: SARIMAX(0, 1, 1) Log Likelihood 299.109
Date: Wed, 22 Oct 2025 AIC -594.218
Time: 21:36:49 BIC -587.216
Sample: 01-01-2000 HQIC -591.398
- 06-01-2020
Covariance Type: opg
==============================================================================
coef std err z P>|z| [0.025 0.975]
------------------------------------------------------------------------------
ma.L1 0.1470 0.055 2.658 0.008 0.039 0.255
sigma2 0.0051 0.000 15.077 0.000 0.004 0.006
===================================================================================
Ljung-Box (L1) (Q): 0.03 Jarque-Bera (JB): 37.44
Prob(Q): 0.86 Prob(JB): 0.00
Heteroskedasticity (H): 1.18 Skew: 0.42
Prob(H) (two-sided): 0.46 Kurtosis: 4.72
===================================================================================
Warnings:
[1] Covariance matrix calculated using the outer product of gradients (complex-step).
1. Independencia temporal (Ljung–Box test)
Prob(Q) = 0.86 → valor p > 0.05
⇒ No se rechaza la hipótesis nula de independencia.
Esto significa que los residuales no presentan autocorrelación significativa,es decir, el modelo ha capturado correctamente la estructura temporal de la serie.
En otras palabras, no queda información predecible en los errores: los residuales se comportan como ruido blanco.
2. Varianza constante (Heteroskedasticity test)
Prob(H) = 0.46 → valor p > 0.05
⇒ No se rechaza la hipótesis nula de homocedasticidad.
Esto indica que la varianza de los residuales es constante en el tiempo,por lo tanto, no hay evidencia de heterocedasticidad.
3. Normalidad (Jarque–Bera, Skew y Kurtosis)
Prob(JB) = 0.00 → valor p < 0.05
⇒ Se rechaza la hipótesis nula de normalidad.
Esto indica que los residuales no siguen una distribución normal exacta.
Sin embargo, los parámetros de forma ayudan a interpretar el tipo de desviación:
Skew = 0.42 → ligera asimetría positiva: la distribución tiene una cola algo más larga a la derecha.
Kurtosis = 4.72 → valor mayor que 3, indicando colas pesadas o leptocurtosis, es decir, mayor presencia de valores extremos que en una normal.
4. Conclusión general
Los residuales son independientes (sin autocorrelación).
La varianza es constante (homocedasticidad).
Distribución no perfectamente normal, aunque con desviaciones leves (colas algo más pesadas y ligera asimetría).
En conjunto, los residuales cumplen las condiciones clave para el uso del modelo en pronóstico:
No hay correlación remanente ni problemas de varianza.
Las desviaciones leves de normalidad no invalidan el modelo, aunque podrían afectar ligeramente los intervalos de confianza.
Interpretación
El modelo está correctamente especificado desde el punto de vista temporal y de estabilidad de varianza.
Los errores se comportan como ruido blanco con forma aproximadamente normal,
lo que permite usar el modelo con confianza para pronósticos fuera de la muestra.
Solo se recomienda cautela al interpretar los intervalos de predicción,ya que la presencia de colas pesadas (Kurtosis > 4) puede subestimar la probabilidad de eventos extremos.
¿Qué significa que “el modelo ha capturado correctamente la estructura temporal”?
Cuando se dice que “ha capturado correctamente la estructura temporal” significa que:
- Los residuales (errores) ya no contienen correlación con el pasado,es decir, no hay patrón temporal remanente.
En otras palabras, el modelo extrajo toda la información temporal disponible de la serie.
Esto se verifica porque:
- El test Ljung–Box tiene un valor p > 0.05,indicando que los residuales son independientes (no autocorrelacionados).
- En los gráficos de ACF y PACF, todas las barras están dentro de las bandas de significancia,lo que confirma que no quedan rezagos significativos.
- Por tanto, lo que queda en los residuales es ruido blanco puro:valores aleatorios con media cero, varianza constante y sin estructura temporal.
Ejemplo
- Si el modelo no capturara bien la estructura temporal,verías que los residuales presentan autocorrelación,o que el test de Ljung–Box resulta significativo (p < 0.05).En ese caso, habría información del pasado que el modelo no aprovechó.
- En cambio, si el modelo sí la captura correctamente,los residuales se distribuyen aleatoriamente a lo largo del tiempo y no se parecen en nada a la serie original.
Conclusión
Decir que “el modelo ha capturado correctamente la estructura temporal” significa quetoda la información dependiente del tiempo presente en la serie ya está explicada por el modelo, y los residuales son simplemente ruido blanco.
En términos prácticos, esto garantiza que el modelo puede utilizarse para pronóstico,porque las dependencias temporales ya fueron incorporadas en su estructura (AR, MA, SAR, etc.).
Ajuste y pronóstico en la serie original:#
###### Pronóstico dentro de la muestra (train) ######
fitted_values = results.fittedvalues
conf_int_train = results.get_prediction().conf_int(alpha=0.05) # Intervalo de confianza del 95%
# Alinear por si el índice de train y fitted_values difieren en los primeros p rezagos
fitted_values = fitted_values.reindex(train.index)
###### Pronóstico fuera de la muestra (test) #####
current_results = results # Modelo ajustado
forecasted_test = []
lower_ci_test = []
upper_ci_test = []
for i in range(len(test)):
forecaster = current_results.get_forecast(steps=1) # Un pronóstico hacia adelante
forecast_mean_test = forecaster.predicted_mean.iloc[0] # Media del pronóstico
ci_i_test = forecaster.conf_int(alpha=0.05).iloc[0] # Intervalo de confianza del 95%
forecasted_test.append(forecast_mean_test)
lower_ci_test.append(ci_i_test.iloc[0]) # límite inferior
upper_ci_test.append(ci_i_test.iloc[1]) # límite superior
# Actualiza el estado con el valor real (método recursivo)
current_results = current_results.append(endog=[test.iloc[i]], refit=False)
forecasted_test = pd.Series(forecasted_test, index=test.index, name='forecast_test')
lower_ci_test = pd.Series(lower_ci_test, index=test.index, name='lower_test')
upper_ci_test = pd.Series(upper_ci_test, index=test.index, name='upper_test')
###### Pronóstico fuera de la muestra: futuro #####
n_forecast = 5 # Pronóstico para 12 meses
# Actualiza el estado con el dataset de test
current_results = results.append(endog=test, refit=False)
forecasting = []
lower_ci = []
upper_ci = []
for i in range(n_forecast):
forecaster = current_results.get_forecast(steps=1) # Un pronóstico hacia adelante
forecast_mean = forecaster.predicted_mean.iloc[0] # Media del pronóstico
ci_i = forecaster.conf_int(alpha=0.05).iloc[0] # Intervalo de confianza del 95%
forecasting.append(forecast_mean)
lower_ci.append(ci_i.iloc[0]) # límite inferior
upper_ci.append(ci_i.iloc[1]) # límite superior
# Alimenta el modelo con el valor pronosticado (pronóstico puro hacia adelante)
current_results = current_results.append(endog=[forecast_mean], refit=False)
# Fechas futuras (mensuales inicio de mes)
last_date = test.index[-1]
future_dates = pd.date_range(start=last_date + pd.offsets.MonthBegin(1),
periods=n_forecast, freq='MS')
# Asegura Series con índice de fechas
forecasting = pd.Series(forecasting, index=future_dates, name='forecast')
lower_ci = pd.Series(lower_ci, index=future_dates, name='lower')
upper_ci = pd.Series(upper_ci, index=future_dates, name='upper')
# Inversa de la transformación - SARIMAX devuelve automáticamente la diferenciación
y_pred_train = np.exp(fitted_values)
y_pred_test = np.exp(forecasted_test)
forcasting_orig = np.exp(forecasting) # pronóstico futuro
# Intervalos de confianza
lower_bt = np.exp(lower_ci)
upper_bt = np.exp(upper_ci)
# Graficar sobre la serie original
plt.figure(figsize=(12,6))
# Serie original
plt.plot(serie[1:], label='Precio de electricidad', color='black')
# Ajuste en train
plt.plot(y_pred_train[1:], label='Ajuste en train', color='tab:blue')
# Ajuste en test
plt.plot(y_pred_test, label='Pronóstico en test', color='tab:green')
# Pronóstico futuro + IC
plt.plot(forcasting_orig, label='Pronóstico futuro', color='tab:red', linestyle='--')
plt.fill_between(future_dates, lower_bt.values, upper_bt.values, color='tab:red', alpha=0.2, label='IC 95%')
plt.title('Ajuste y pronóstico')
plt.xlabel('Tiempo')
plt.ylabel('Valor')
plt.legend()
plt.tight_layout()
plt.show()

Análisis de residuales#
y_pred = y_pred_train[1:]
y_real = serie[1:split]
plt.figure(figsize=(20,6))
# Serie real
plt.plot(y_real, label='Serie real', color='black', linewidth=2)
# Valores ajustados o predichos
plt.plot(y_pred, label='Ajuste del modelo', color='blue', linewidth=2, alpha=0.8)
plt.title("Ajuste sobre Train", fontsize=12)
plt.xlabel("Tiempo")
plt.ylabel("Valor")
plt.legend()
plt.grid(alpha=0.3)
plt.tight_layout()
plt.show()
# Extraer residuales
residuals = results.resid
print(residuals.head())
Fecha
2000-01-01 12.824968
2000-02-01 -0.047077
2000-03-01 0.022787
2000-04-01 -0.038623
2000-05-01 0.023394
Freq: MS, dtype: float64
residuals = results.resid[1:]
Note que el primer residual tiene un valor muy desfasado, trabajaremos sin ese primer valor
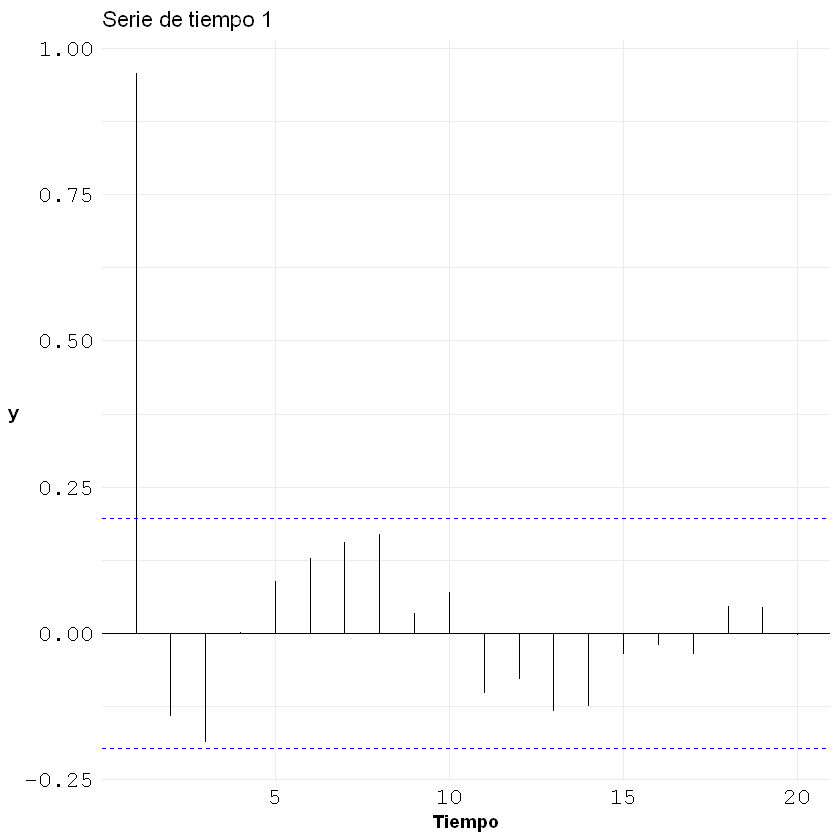
Gráfico de residuales en el tiempo#
# Gráfico en el tiempo
plt.figure(figsize=(11,4))
plt.scatter(residuals.index, residuals, color="darkblue")
plt.axhline(0, ls="--", color="black")
plt.title("Residuales en el tiempo")
plt.xlabel("Tiempo")
plt.ylabel("Residual")
plt.tight_layout()
plt.show()
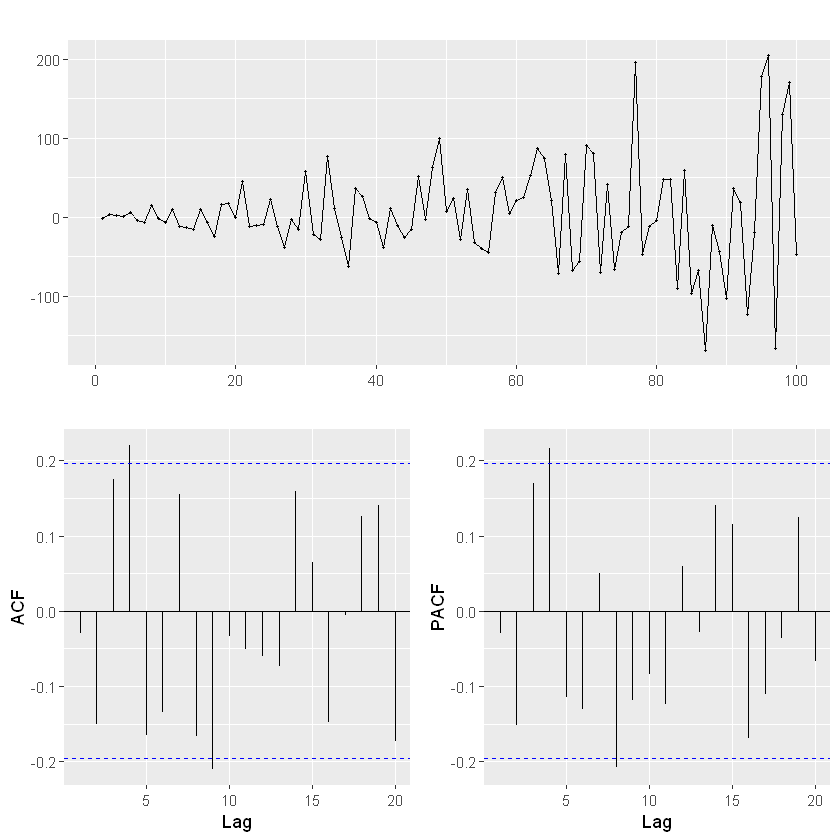
ACF y PACF de los residuales#
import statsmodels.api as sm
fig, axes = plt.subplots(1, 2, figsize=(12,4))
# Gráfico ACF
sm.graphics.tsa.plot_acf(residuals, lags=20, ax=axes[0], zero=False, color='navy')
axes[0].set_title("ACF de los residuales")
axes[0].set_xlabel("Rezagos")
axes[0].set_ylabel("Autocorrelación")
# Gráfico PACF
sm.graphics.tsa.plot_pacf(residuals, lags=20, ax=axes[1], zero=False, color='navy')
axes[1].set_title("PACF de los residuales")
axes[1].set_xlabel("Rezagos")
axes[1].set_ylabel("Autocorrelación parcial")
plt.tight_layout()
plt.show()
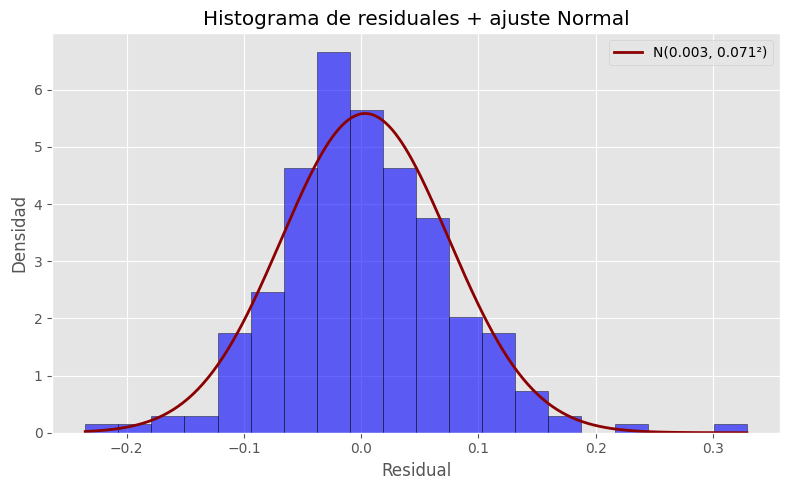
Histograma de los residuales#
# Histograma de residuales con ajuste Normal
from scipy.stats import norm
# Parámetros de la normal ajustada (MLE)
mu = residuals.mean()
sigma = residuals.std(ddof=1)
# Rango para la curva teórica
x = np.linspace(residuals.min(), residuals.max(), 400)
pdf = norm.pdf(x, loc=mu, scale=sigma)
plt.figure(figsize=(8,5))
plt.hist(residuals, bins="auto", density=True, alpha=0.6, edgecolor="k", color="blue")
plt.plot(x, pdf, lw=2, label=f"N({mu:.3f}, {sigma:.3f}²)", color="darkred")
plt.title("Histograma de residuales + ajuste Normal")
plt.xlabel("Residual")
plt.ylabel("Densidad")
plt.legend()
plt.tight_layout()
plt.show()
QQ-plot de los residuales#
plt.figure(figsize=(6,6))
sm.qqplot(residuals, line='45', fit=True)
plt.title("Q-Q Plot de los residuales")
plt.xlabel("Cuantiles teóricos (Normal)")
plt.ylabel("Cuantiles de los residuales")
plt.tight_layout()
plt.show()
<Figure size 600x600 with 0 Axes>
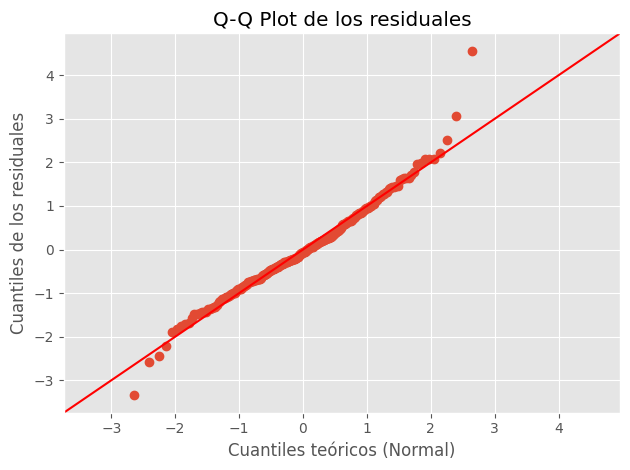
Interpretación:
Si los puntos se alinean cerca de la línea diagonal (45°), los residuales siguen una distribución aproximadamente normal.
Si los puntos se desvían sistemáticamente (por ejemplo, curvándose hacia arriba o hacia abajo), los residuales no son normales:
Desviaciones en los extremos → colas más pesadas o más ligeras.
Curvatura en el centro → asimetría (sesgo a la derecha o izquierda)
Gráfico de valores predichos vs. valores reales#
plt.figure(figsize=(6,6))
plt.scatter(y_real, y_pred, color='blue', alpha=0.6, edgecolor='k')
# Línea de identidad (y = x)
min_val = min(y_real.min().values, y_pred.min())
max_val = max(y_real.max().values, y_pred.max())
plt.plot([min_val, max_val], [min_val, max_val], color='black', lw=2)
plt.title("Valores predichos vs. valores reales", fontsize=12)
plt.xlabel("Valores reales")
plt.ylabel("Valores predichos")
plt.axis("equal") # asegura proporciones iguales para la diagonal
plt.grid(alpha=0.3)
plt.tight_layout()
plt.show()
Interpretación análisis de los residuales:#
Los residuales del modelo se analizaron mediante estadísticos de diagnóstico y herramientas gráficas.
Los resultados fueron los siguientes:
1. Independencia temporal (Ljung–Box test)
El valor de Prob(Q) = 0.86 es muy superior a 0.05, por lo que no se rechaza la hipótesis nula de independencia.
Esto indica que no hay autocorrelación significativa en los residuales, es decir, el modelo ha capturado adecuadamente la estructura temporal de la serie.
2. Varianza constante (Heteroskedasticity test)
El valor Prob(H) = 0.46 tampoco es significativo (p > 0.05), lo que sugiere que la varianza de los residuales es constante a lo largo del tiempo.
Visualmente, los residuales se mantienen con amplitud similar alrededor de cero, sin ensanchamientos o estrechamientos sistemáticos.
Esto confirma que no hay heterocedasticidad en el modelo.
3. Normalidad (Jarque–Bera, Skew y Kurtosis)
El estadístico JB = 37.44 con p = 0.00 indica que se rechaza la hipótesis nula de normalidad.
Sin embargo, al examinar el histograma y el Q-Q plot, se observa que:
- Los residuales están centrados en cero y el histograma tiene forma de campana simétrica,con solo colas ligeramente más pesadas a ambos lados.
El Q-Q plot confirma esta observación:
los puntos siguen aproximadamente la línea de 45°,
con ligeras desviaciones en los extremos (izquierda por debajo de la línea, derecha por encima).
En conjunto, esto indica que la distribución es casi normal, con leve exceso de curtosis (Kurtosis = 4.72) y una asimetría leve hacia la derecha (Skew = 0.42).
4. Presencia de valores atípicos
Solo se identifican tres residuales claramente extremos respecto al resto.
Estos no alteran el patrón general, por lo que se consideran atípicos aislados y no indicadores de un problema estructural del modelo.
Conclusión general
Los residuales están centrados en cero, no autocorrelacionados (ACF y PACF dentro de bandas), y con varianza constante.
Presentan forma aproximadamente normal, con colas ligeramente más pesadas, lo cual es aceptable para propósitos de pronóstico.
- En términos generales, el modelo se considera bien especificado, y los residuales se comportan casi como ruido blanco,por lo que puede usarse con confianza para generar pronósticos.