Árboles de decisión para series de tiempo#
import numpy as np
import pandas as pd
from sklearn.tree import DecisionTreeRegressor
from sklearn.model_selection import train_test_split
from sklearn.metrics import mean_absolute_error, mean_squared_error, r2_score
import matplotlib.pyplot as plt
import scipy.stats as stats
from sklearn.model_selection import RandomizedSearchCV
import warnings
# Suprimir todas las advertencias
warnings.filterwarnings("ignore")
# Cargar los datos omitiendo la primera fila como encabezado y asignando nombres a las columnas
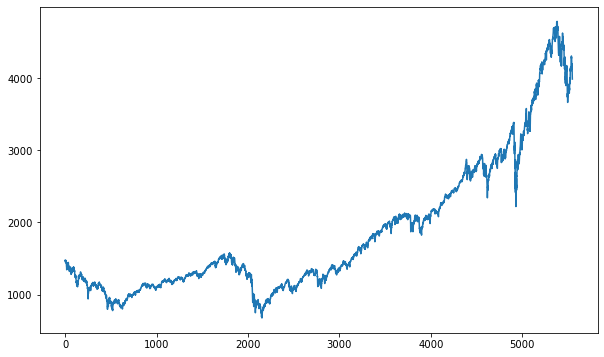
data = pd.read_csv("../Irradiance_mensual.csv", skiprows=1, header=None, names=['Fecha', 'Irradiancia'])
# Convertir la columna 'Fecha' a datetime
data['Fecha'] = pd.to_datetime(data['Fecha'], format='%Y-%m')
# Set 'Fecha' as the index
data.set_index('Fecha', inplace=True)
# Cantidad de datos
n = data.shape[0]
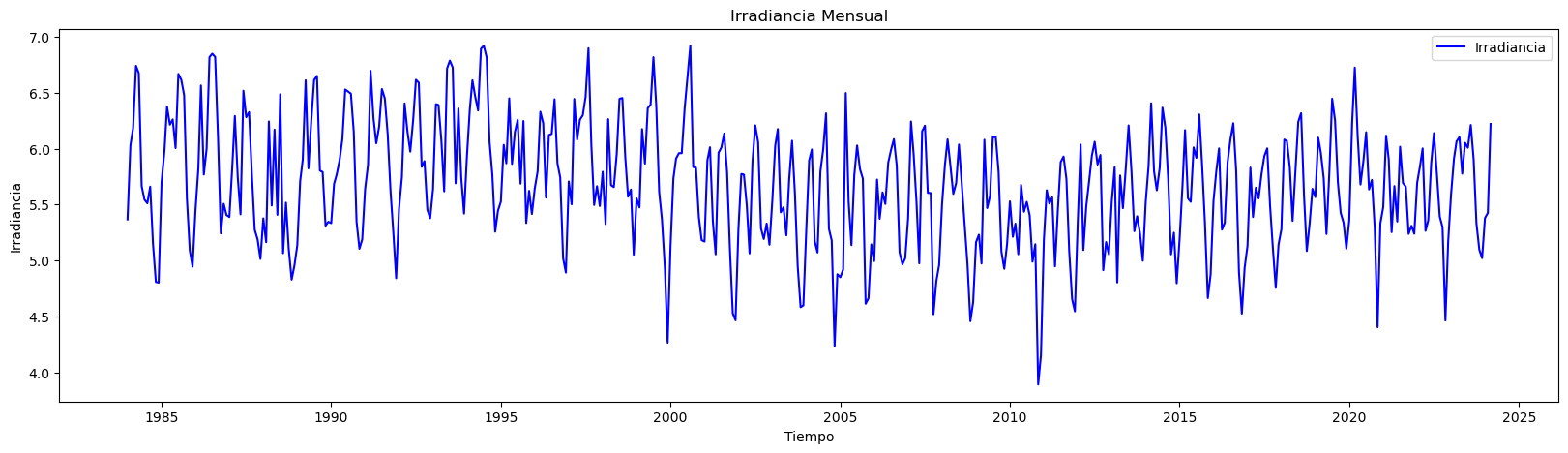
print("Cantidad de datos:", n)
plt.figure(figsize=(20, 5)) # Establecer el tamaño del gráfico
plt.plot(data.index, data['Irradiancia'], label='Irradiancia', color='blue') # Dibujar los datos reales
plt.title('Irradiancia Mensual') # Título del gráfico
plt.xlabel('Tiempo') # Etiqueta del eje X
plt.ylabel('Irradiancia') # Etiqueta del eje Y
plt.legend() # Añadir leyenda para identificar las líneas
plt.show()
Cantidad de datos: 483
Conjunto de Train y Test#
# Definir características (X) y objetivo (y)
X = data[["Irradiancia"]] # Usar la irradiancia como característica
y = data["Irradiancia"] # Usar la irradiancia también como objetivo
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, shuffle=False)
Árboles de decisión para regresión:#
# Crear el modelo de árbol de decisión
tree_regressor = DecisionTreeRegressor(random_state=34)
# Entrenar el modelo en el conjunto de entrenamiento
tree_regressor.fit(X_train, y_train)
DecisionTreeRegressor(random_state=34)
Evaluación del modelo:#
Sobre el conjunto de Train:
# Predicciones en el conjunto de prueba
y_pred_train = tree_regressor.predict(X_train)
# Calcular MAE
mae_train = mean_absolute_error(y_train, y_pred_train)
# Calcular MSE
mse_train = mean_squared_error(y_train, y_pred_train)
# Calcular RMSE
rmse_train = np.sqrt(mse_train)
# Calcular R² Score
r2_train = r2_score(y_train, y_pred_train)
# Mostar métricas
print("Métricas en el conjunto de entrenamiento:")
print("Mean Absolute Error (MAE):", mae_train)
print("Mean Squared Error (MSE):", mse_train)
print("Root Mean Squared Error (RMSE):", rmse_train)
print("R² Score:", r2_train)
Métricas en el conjunto de entrenamiento:
Mean Absolute Error (MAE): 0.0
Mean Squared Error (MSE): 0.0
Root Mean Squared Error (RMSE): 0.0
R² Score: 1.0
Sobre el conjunto de Test:
# Predicciones en el conjunto de prueba
y_pred = tree_regressor.predict(X_test)
# Calcular MAE
mae = mean_absolute_error(y_test, y_pred)
# Calcular MSE
mse = mean_squared_error(y_test, y_pred)
# Calcular RMSE
rmse = np.sqrt(mse)
# Calcular R² Score
r2 = r2_score(y_test, y_pred)
# Mostrar las métricas
print("Métricas en el conjunto de entrenamiento:")
print(f"Mean Absolute Error (MAE): {mae}")
print(f"Mean Squared Error (MSE): {mse}")
print(f"Root Mean Squared Error (RMSE): {rmse}")
print(f"R² Score: {r2}")
Métricas en el conjunto de entrenamiento:
Mean Absolute Error (MAE): 0.003318427500582964
Mean Squared Error (MSE): 5.726579999023996e-05
Root Mean Squared Error (RMSE): 0.007567416995926679
R² Score: 0.9997066910209671
# Gráfica de resultados
plt.figure(figsize=(14, 7))
plt.plot(y_test.index, y_test.values, label="Test", color="green")
plt.plot(y_test.index, y_pred, label="Predicted", linestyle="--", color="red")
plt.title("Ajuste modelo árboles de decisión")
plt.xlabel("Fecha")
plt.ylabel("Irradiancia")
plt.legend()
plt.show()
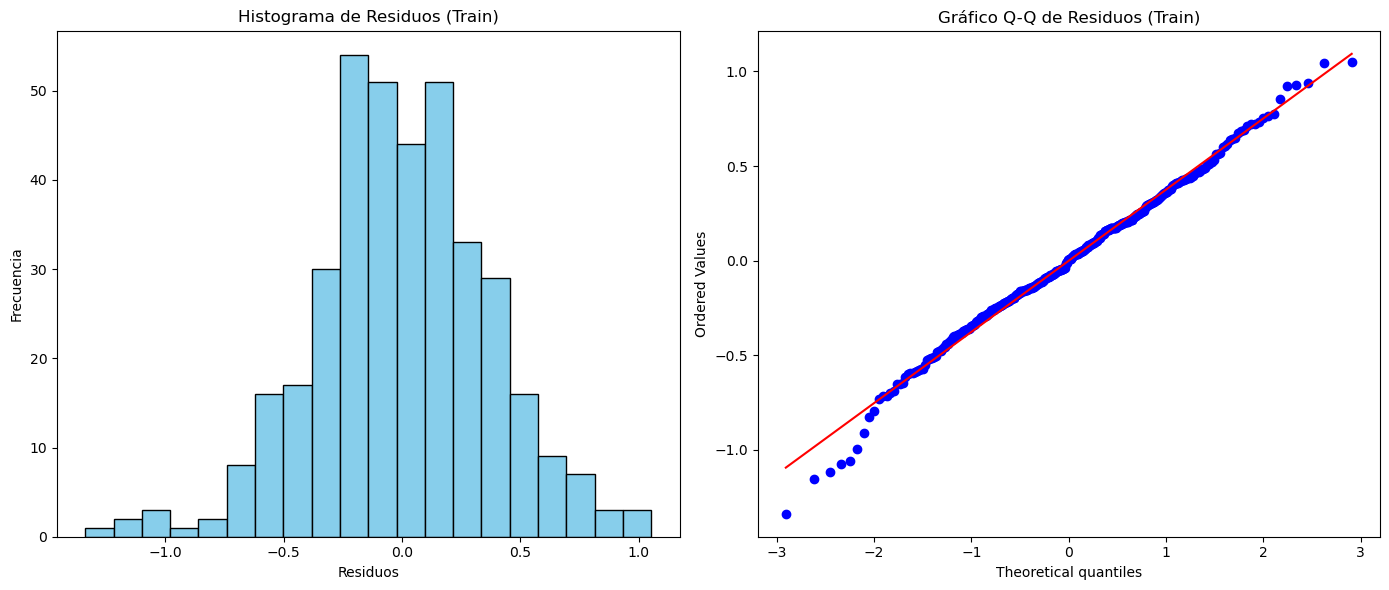
Análisis de residuales:#
# Hacer predicciones en el conjunto de entrenamiento
y_pred_train = tree_regressor.predict(X_train)
# Calcular los residuales sobre el conjunto de entrenamiento
residuals_train = y_train - y_pred_train
# Configuración de la figura para los subplots
fig, axs = plt.subplots(nrows=1, ncols=2, figsize=(14, 6))
# Gráfico de valores predichos vs. valores reales
axs[0].scatter(y_pred_train, y_train, color="blue", alpha=0.5)
axs[0].plot(
[y_train.min(), y_train.max()], [y_train.min(), y_train.max()], "k--", lw=2
) # Línea diagonal ideal
axs[0].set_title("Valores Reales vs. Valores Predichos (Train)")
axs[0].set_xlabel("Valores Predichos")
axs[0].set_ylabel("Valores Reales")
# Gráfico de residuales
axs[1].scatter(y_train.index, residuals_train, color="purple", alpha=0.3)
axs[1].axhline(y=0, color="black", linestyle="--") # Línea en y=0 para referencia
axs[1].set_title("Gráfico de Residuales (Train)")
axs[1].set_xlabel("Tiempo")
axs[1].set_ylabel("Residuales")
# Mejorar el layout para evitar solapamientos
plt.tight_layout()
# Mostrar la figura
plt.show()
# Visualización del histograma de los residuos
plt.figure(figsize=(14, 6))
plt.subplot(1, 2, 1)
plt.hist(residuals_train, bins=20, color="skyblue", edgecolor="black")
plt.title("Histograma de Residuos (Train)")
plt.xlabel("Residuos")
plt.ylabel("Frecuencia")
# Visualización del gráfico Q-Q de los residuos
plt.subplot(1, 2, 2)
stats.probplot(residuals_train, dist="norm", plot=plt)
plt.title("Gráfico Q-Q de Residuos (Train)")
# Ajustar el diseño de la figura
plt.tight_layout()
# Mostrar la figura
plt.show()
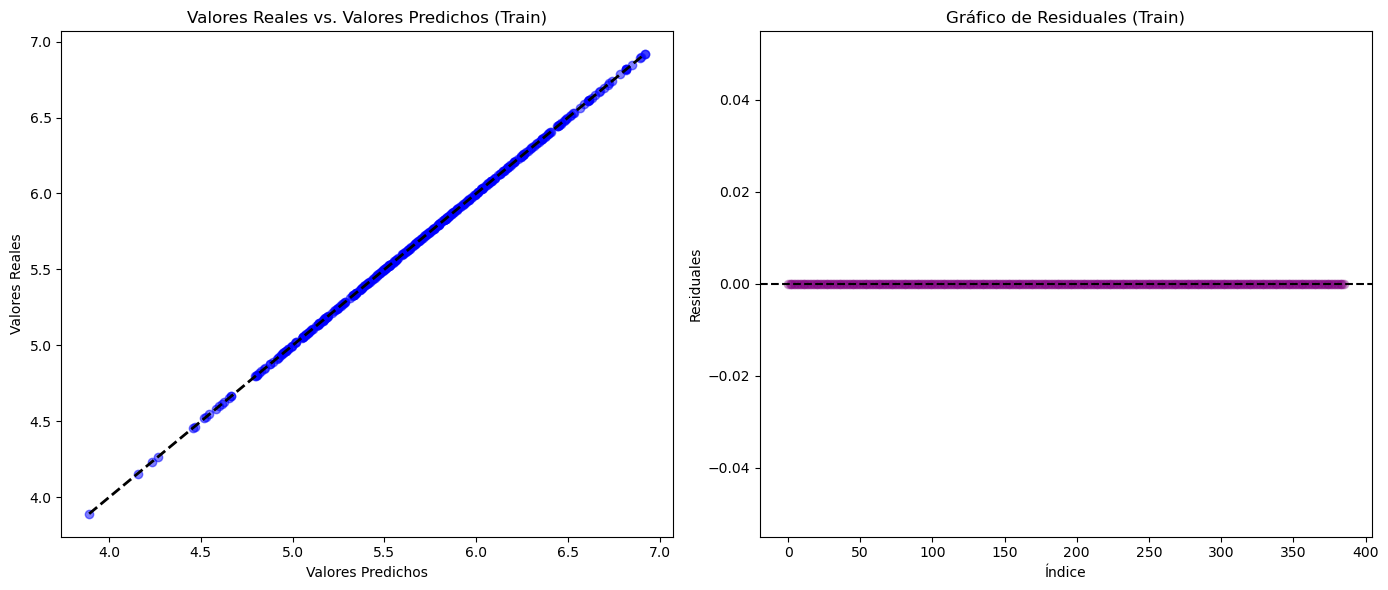
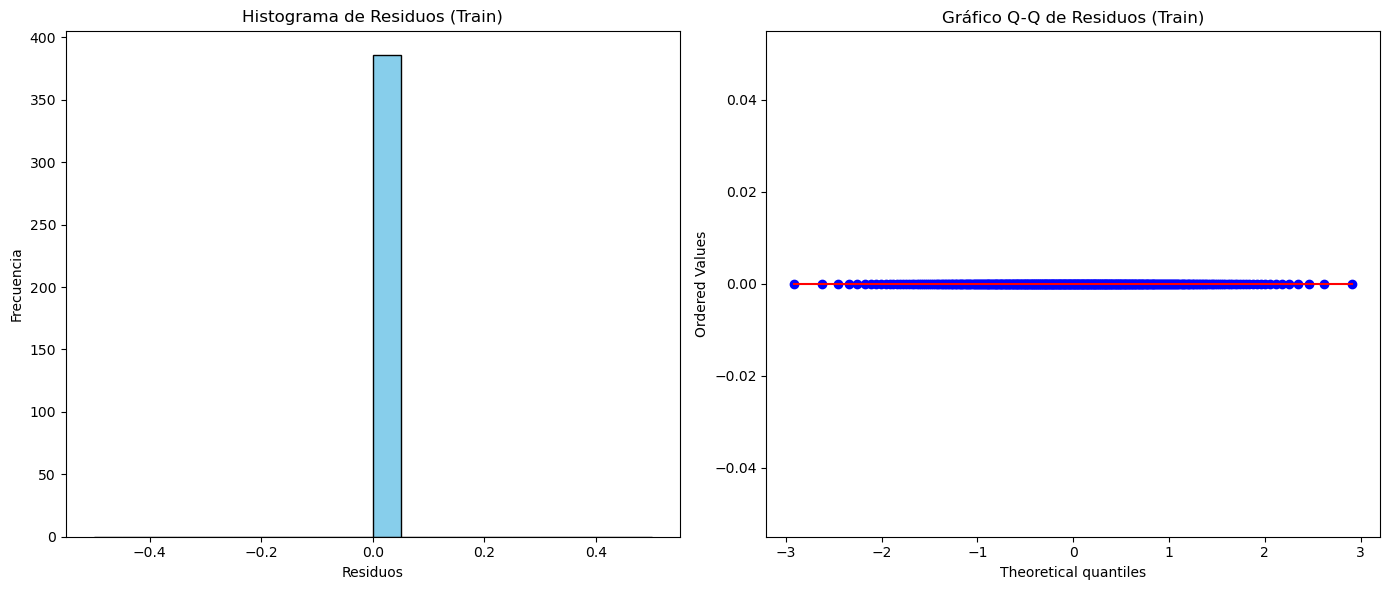
Predicciones fuera de la muestra:#
# Tomar el último valor de X_test como punto de partida para las predicciones fuera de muestra
last_X = X_test.iloc[-1].values.reshape(1, -1)
# Número de pasos adelante para predecir
n_steps_ahead = 24
# Array para almacenar las predicciones fuera de muestra
predictions_out_of_sample = []
for _ in range(n_steps_ahead):
# Hacer la predicción usando el último valor de X
pred = tree_regressor.predict(last_X)
# Guardar la predicción
predictions_out_of_sample.append(pred[0])
# Crear la nueva entrada para la siguiente predicción
# Aquí se utiliza la predicción actual como la siguiente entrada
last_X = np.array(pred).reshape(1, -1)
# Crear un rango de fechas para las predicciones fuera de muestra
dates_out_of_sample = pd.date_range(
start=y_test.index[-1], periods=n_steps_ahead + 1, freq="M"
)[1:]
# Graficar las predicciones fuera de muestra
plt.figure(figsize=(10, 6))
plt.plot(y_test.index, y_test, label="Datos Reales (Test)")
plt.plot(
dates_out_of_sample,
predictions_out_of_sample,
label="Predicciones Fuera de Muestra",
color="red",
linestyle="--",
)
plt.title("Predicciones Fuera de Muestra con Árbol de Decisión")
plt.xlabel("Fecha")
plt.ylabel("Valores Predichos")
plt.legend()
plt.show()
Optimización de Hiperparámetros:#
# Crear el modelo de árbol de decisión
tree_regressor = DecisionTreeRegressor(random_state=34)
# Definir el espacio de hiperparámetros
param_dist = {
"max_depth": list(np.arange(1, 20)), # Profundidad máxima del árbol
"min_samples_split": np.arange(2, 20), # Mínimo número de muestras para dividir un nodo
"min_samples_leaf": np.arange(5, 20), # Mínimo número de muestras en una hoja
"max_features": [None, "auto", "sqrt", "log2"], # Número de características a considerar para la mejor división
"max_leaf_nodes": [None] + list(np.arange(2, 100)), # Número máximo de nodos hoja
}
# Configurar RandomizedSearchCV
random_search = RandomizedSearchCV(
estimator=tree_regressor,
param_distributions=param_dist,
n_iter=300, # Número de combinaciones aleatorias a evaluar
scoring="neg_mean_squared_error", # Usar MSE negativo como métrica de scoring
cv=5, # Validación cruzada con 5 pliegues
verbose=2,
random_state=34,
n_jobs=-1, # Usar todos los núcleos disponibles
)
# Ajustar RandomizedSearchCV al conjunto de entrenamiento
random_search.fit(X_train, y_train)
Fitting 5 folds for each of 300 candidates, totalling 1500 fits
RandomizedSearchCV(cv=5, estimator=DecisionTreeRegressor(random_state=34),
n_iter=300, n_jobs=-1,
param_distributions={'max_depth': [1, 2, 3, 4, 5, 6, 7, 8, 9,
10, 11, 12, 13, 14, 15,
16, 17, 18, 19],
'max_features': [None, 'auto', 'sqrt',
'log2'],
'max_leaf_nodes': [None, 2, 3, 4, 5, 6,
7, 8, 9, 10, 11, 12,
13, 14, 15, 16, 17,
18, 19, 20, 21, 22,
23, 24, 25, 26, 27,
28, 29, 30, ...],
'min_samples_leaf': array([ 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19]),
'min_samples_split': array([ 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18,
19])},
random_state=34, scoring='neg_mean_squared_error',
verbose=2)
# Obtener los mejores hiperparámetros
best_params = random_search.best_params_
best_score = random_search.best_score_
print(f"Mejores hiperparámetros: {best_params}")
Mejores hiperparámetros: {'min_samples_split': 3, 'min_samples_leaf': 5, 'max_leaf_nodes': 83, 'max_features': 'sqrt', 'max_depth': 14}
# Mejor modelo encontrado:
best_model = random_search.best_estimator_
# Predicciones en el conjunto de prueba
y_pred_train = best_model.predict(X_train)
# Calcular MAE
mae_train = mean_absolute_error(y_train, y_pred_train)
# Calcular MSE
mse_train = mean_squared_error(y_train, y_pred_train)
# Calcular RMSE
rmse_train = np.sqrt(mse_train)
# Calcular R² Score
r2_train = r2_score(y_train, y_pred_train)
# Mostar métricas
print("Métricas en el conjunto de entrenamiento:")
print("Mean Absolute Error (MAE):", mae_train)
print("Mean Squared Error (MSE):", mse_train)
print("Root Mean Squared Error (RMSE):", rmse_train)
print("R² Score:", r2_train)
Métricas en el conjunto de entrenamiento:
Mean Absolute Error (MAE): 0.010106275359833619
Mean Squared Error (MSE): 0.0005711430968164161
Root Mean Squared Error (RMSE): 0.023898600310821888
R² Score: 0.9981737629051306
# Predicciones en el conjunto de prueba
y_pred = best_model.predict(X_test)
# Calcular MAE
mae = mean_absolute_error(y_test, y_pred)
# Calcular MSE
mse = mean_squared_error(y_test, y_pred)
# Calcular RMSE
rmse = np.sqrt(mse)
# Calcular R² Score
r2 = r2_score(y_test, y_pred)
# Mostrar las métricas
print("Métricas en el conjunto de entrenamiento:")
print(f"Mean Absolute Error (MAE): {mae}")
print(f"Mean Squared Error (MSE): {mse}")
print(f"Root Mean Squared Error (RMSE): {rmse}")
print(f"R² Score: {r2}")
Métricas en el conjunto de entrenamiento:
Mean Absolute Error (MAE): 0.011398115305865372
Mean Squared Error (MSE): 0.00060460039986064
Root Mean Squared Error (RMSE): 0.02458862338278904
R² Score: 0.996903304834016
# Gráfica de resultados
plt.figure(figsize=(14, 7))
plt.plot(y_test.index, y_test.values, label="Test", color="green")
plt.plot(y_test.index, y_pred, label="Predicted", linestyle="--", color="red")
plt.title("Ajuste modelo árboles de decisión")
plt.xlabel("Fecha")
plt.ylabel("Irradiancia")
plt.legend()
plt.show()
Residuales:
# Hacer predicciones en el conjunto de entrenamiento
y_pred_train = best_model.predict(X_train)
# Calcular los residuales sobre el conjunto de entrenamiento
residuals_train = y_train - y_pred_train
# Configuración de la figura para los subplots
fig, axs = plt.subplots(nrows=1, ncols=2, figsize=(14, 6))
# Gráfico de valores predichos vs. valores reales
axs[0].scatter(y_pred_train, y_train, color="blue", alpha=0.5)
axs[0].plot(
[y_train.min(), y_train.max()], [y_train.min(), y_train.max()], "k--", lw=2
) # Línea diagonal ideal
axs[0].set_title("Valores Reales vs. Valores Predichos (Train)")
axs[0].set_xlabel("Valores Predichos")
axs[0].set_ylabel("Valores Reales")
# Gráfico de residuales
axs[1].scatter(y_train.index, residuals_train, color="purple", alpha=0.3)
axs[1].axhline(y=0, color="black", linestyle="--") # Línea en y=0 para referencia
axs[1].set_title("Gráfico de Residuales (Train)")
axs[1].set_xlabel("Tiempo")
axs[1].set_ylabel("Residuales")
# Mejorar el layout para evitar solapamientos
plt.tight_layout()
# Mostrar la figura
plt.show()
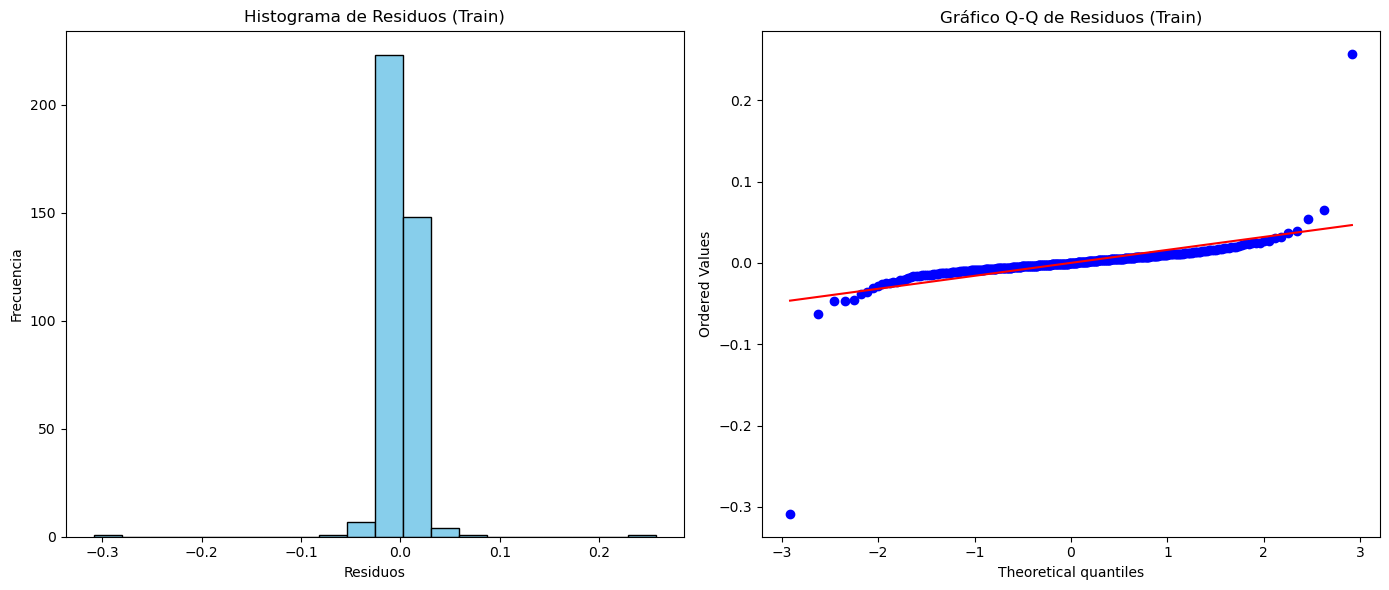
# Visualización del histograma de los residuos
plt.figure(figsize=(14, 6))
plt.subplot(1, 2, 1)
plt.hist(residuals_train, bins=20, color="skyblue", edgecolor="black")
plt.title("Histograma de Residuos (Train)")
plt.xlabel("Residuos")
plt.ylabel("Frecuencia")
# Visualización del gráfico Q-Q de los residuos
plt.subplot(1, 2, 2)
stats.probplot(residuals_train, dist="norm", plot=plt)
plt.title("Gráfico Q-Q de Residuos (Train)")
# Ajustar el diseño de la figura
plt.tight_layout()
# Mostrar la figura
plt.show()
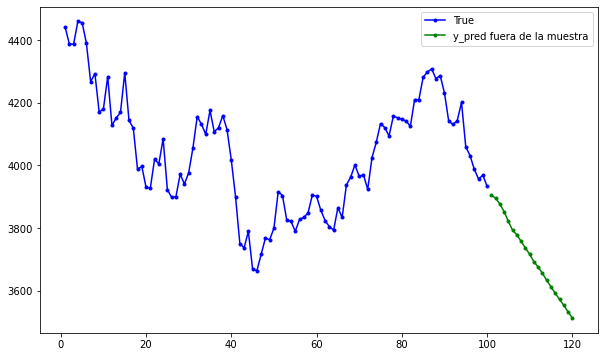
Predicciones fuera de la muestra:#
# Tomar el último valor de X_test como punto de partida para las predicciones fuera de muestra
last_X = X_test.iloc[-1].values.reshape(1, -1)
# Número de pasos adelante para predecir
n_steps_ahead = 12 * 5
# Array para almacenar las predicciones fuera de muestra
predictions_out_of_sample = []
for _ in range(n_steps_ahead):
# Hacer la predicción usando el último valor de X
pred = best_model.predict(last_X)
# Guardar la predicción
predictions_out_of_sample.append(pred[0])
# Crear la nueva entrada para la siguiente predicción
# Aquí se utiliza la predicción actual como la siguiente entrada
last_X = np.array(pred).reshape(1, -1)
# Crear un rango de fechas para las predicciones fuera de muestra
dates_out_of_sample = pd.date_range(
start=y_test.index[-1], periods=n_steps_ahead + 1, freq="M"
)[1:]
# Graficar las predicciones fuera de muestra
plt.figure(figsize=(10, 6))
plt.plot(y_test.index, y_test, label="Datos Reales (Test)")
plt.plot(
dates_out_of_sample,
predictions_out_of_sample,
label="Predicciones Fuera de Muestra",
color="red",
linestyle="--",
)
plt.title("Predicciones Fuera de Muestra con Árbol de Decisión")
plt.xlabel("Fecha")
plt.ylabel("Valores Predichos")
plt.legend()
plt.show()
Lags de la serie de tiempo como variables de entrada:#
Los árboles de decisión son muy sensibles a los datos; si se modifican los lags, los resultados pueden variar significativamente.
# Crear características de lags
n_lags = 7 # Número de lags que deseas utilizar
data_lags = data.copy()
for lag in range(1, n_lags + 1):
data_lags[f"Irradiancia_Lag_{lag}"] = data["Irradiancia"].shift(lag)
# Eliminar las filas con valores NaN resultantes de los lags
data_lags.dropna(inplace=True)
print(data_lags.head())
Irradiancia Irradiancia_Lag_1 Irradiancia_Lag_2 Fecha
1984-08-01 5.511290 5.544516 5.665000
1984-09-01 5.660333 5.511290 5.544516
1984-10-01 5.150323 5.660333 5.511290
1984-11-01 4.808667 5.150323 5.660333
1984-12-01 4.802258 4.808667 5.150323
Irradiancia_Lag_3 Irradiancia_Lag_4 Irradiancia_Lag_5 Fecha
1984-08-01 6.674194 6.739667 6.182903
1984-09-01 5.665000 6.674194 6.739667
1984-10-01 5.544516 5.665000 6.674194
1984-11-01 5.511290 5.544516 5.665000
1984-12-01 5.660333 5.511290 5.544516
Irradiancia_Lag_6 Irradiancia_Lag_7
Fecha
1984-08-01 6.030690 5.367742
1984-09-01 6.182903 6.030690
1984-10-01 6.739667 6.182903
1984-11-01 6.674194 6.739667
1984-12-01 5.665000 6.674194
# Separar características (X) y la variable objetivo (y)
X_lags = data_lags.drop(columns=["Irradiancia"])
y_lags = data_lags["Irradiancia"]
X_train_lags, X_test_lags, y_train_lags, y_test_lags = train_test_split(
X_lags, y_lags, test_size=0.2, shuffle=False, random_state=34
)
# Crear el modelo de árbol de decisión
tree_regressor_lags = DecisionTreeRegressor(random_state=34)
# Definir el espacio de hiperparámetros
param_dist = {
"max_depth": list(np.arange(1, 20)),
"min_samples_split": np.arange(2, 20),
"min_samples_leaf": np.arange(5, 20),
"max_features": [None, "auto", "sqrt", "log2"],
"max_leaf_nodes": [None] + list(np.arange(2, 100)),
}
# Configurar RandomizedSearchCV
random_search_lags = RandomizedSearchCV(
estimator=tree_regressor_lags,
param_distributions=param_dist,
n_iter=300,
scoring="neg_mean_squared_error",
cv=5,
verbose=2,
random_state=42,
n_jobs=-1,
)
# Ajustar RandomizedSearchCV al conjunto de entrenamiento
random_search_lags.fit(X_train_lags, y_train_lags)
# Mejor modelo encontrado:
best_model_lags = random_search_lags.best_estimator_
# Obtener los mejores hiperparámetros
best_params_lags = random_search_lags.best_params_
print(f"Mejores hiperparámetros: {best_params_lags}")
Fitting 5 folds for each of 300 candidates, totalling 1500 fits
Mejores hiperparámetros: {'min_samples_split': 11, 'min_samples_leaf': 17, 'max_leaf_nodes': 86, 'max_features': None, 'max_depth': 19}
# Predicciones en el conjunto de prueba
y_pred_train = best_model_lags.predict(X_train_lags)
# Calcular MAE
mae_train = mean_absolute_error(y_train_lags, y_pred_train)
# Calcular MSE
mse_train = mean_squared_error(y_train_lags, y_pred_train)
# Calcular RMSE
rmse_train = np.sqrt(mse_train)
# Calcular R² Score
r2_train = r2_score(y_train_lags, y_pred_train)
# Mostar métricas
print("Métricas en el conjunto de entrenamiento:")
print("Mean Absolute Error (MAE):", mae_train)
print("Mean Squared Error (MSE):", mse_train)
print("Root Mean Squared Error (RMSE):", rmse_train)
print("R² Score:", r2_train)
Métricas en el conjunto de entrenamiento:
Mean Absolute Error (MAE): 0.29360416854625876
Mean Squared Error (MSE): 0.14067565958623438
Root Mean Squared Error (RMSE): 0.37506754003277115
R² Score: 0.5479854699681128
# Predicciones en el conjunto de prueba
y_pred_lags = best_model_lags.predict(X_test_lags)
# Calcular MAE
mae = mean_absolute_error(y_test_lags, y_pred_lags)
# Calcular MSE
mse = mean_squared_error(y_test_lags, y_pred_lags)
# Calcular RMSE
rmse = np.sqrt(mse)
# Calcular R² Score
r2 = r2_score(y_test_lags, y_pred_lags)
# Mostrar las métricas
print("Métricas en el conjunto de entrenamiento:")
print(f"Mean Absolute Error (MAE): {mae}")
print(f"Mean Squared Error (MSE): {mse}")
print(f"Root Mean Squared Error (RMSE): {rmse}")
print(f"R² Score: {r2}")
Métricas en el conjunto de entrenamiento:
Mean Absolute Error (MAE): 0.30917753360149003
Mean Squared Error (MSE): 0.16115155223053157
Root Mean Squared Error (RMSE): 0.40143685958134384
R² Score: 0.17695088832056238
# Gráfica de resultados
plt.figure(figsize=(14, 7))
plt.plot(y_test[1:].index, y_test[1:].values, label="Test", color="green")
plt.plot(y_test_lags.index, y_pred_lags, label="Predicted", linestyle="--", color="red")
plt.title("Ajuste modelo árboles de decisión")
plt.xlabel("Fecha")
plt.ylabel("Irradiancia")
plt.legend()
plt.show()
# Preparar el punto de partida (última fila de X_test)
last_X_lags = X_test_lags.iloc[-1].values.reshape(1, -1)
# Número de pasos adelante para predecir
n_steps_ahead = 12 * 5
# Lista para almacenar las predicciones fuera de muestra
predictions_out_of_sample = []
for _ in range(n_steps_ahead):
# Hacer la predicción usando el último valor de X
pred = best_model_lags.predict(last_X_lags)
# Guardar la predicción
predictions_out_of_sample.append(pred[0])
# Crear la nueva entrada para la siguiente predicción utilizando los lags
new_X = np.roll(last_X_lags, shift=-1) # Desplazar valores
new_X[0, -n_lags:] = pred # Actualizar con la nueva predicción
last_X_lags = new_X.reshape(1, -1) # Reajustar la forma
# Crear un rango de fechas para las predicciones fuera de muestra
dates_out_of_sample = pd.date_range(
start=y_test.index[-1], periods=n_steps_ahead + 1, freq="M"
)[1:]
# Graficar las predicciones fuera de muestra
plt.figure(figsize=(10, 6))
plt.plot(y_test.index, y_test, label="Datos Reales (Test)")
plt.plot(
dates_out_of_sample,
predictions_out_of_sample,
label="Predicciones Fuera de Muestra",
color="red",
linestyle="--",
)
plt.title("Predicciones fuera de muestra con árbol de decisión (con Lags)")
plt.xlabel("Fecha")
plt.ylabel("Valores Predichos")
plt.legend()
plt.show()
Residuales:
# Hacer predicciones en el conjunto de entrenamiento con el mejor modelo
y_pred_train = best_model_lags.predict(X_train_lags)
# Calcular los residuales en el conjunto de entrenamiento
residuals_train = y_train_lags - y_pred_train
# Configuración de la figura para los subplots
fig, axs = plt.subplots(nrows=1, ncols=2, figsize=(14, 6))
# Gráfico de valores predichos vs. valores reales
axs[0].scatter(y_pred_train, y_train[n_lags - 1 :], color="blue", alpha=0.5)
axs[0].plot(
[y_train.min(), y_train.max()], [y_train.min(), y_train.max()], "k--", lw=2
) # Línea diagonal ideal
axs[0].set_title("Valores Reales vs. Valores Predichos (Train)")
axs[0].set_xlabel("Valores Predichos")
axs[0].set_ylabel("Valores Reales")
# Gráfico de residuales
axs[1].scatter(y_train.index, residuals_train, color="purple", alpha=0.3)
axs[1].axhline(y=0, color="black", linestyle="--") # Línea en y=0 para referencia
axs[1].set_title("Gráfico de Residuales (Train)")
axs[1].set_xlabel("Tiempo")
axs[1].set_ylabel("Residuales")
# Mejorar el layout para evitar solapamientos
plt.tight_layout()
plt.show()
# Visualización del histograma de los residuos
plt.figure(figsize=(14, 6))
plt.subplot(1, 2, 1)
plt.hist(residuals_train, bins=20, color="skyblue", edgecolor="black")
plt.title("Histograma de Residuos (Train)")
plt.xlabel("Residuos")
plt.ylabel("Frecuencia")
# Visualización del gráfico Q-Q de los residuos
plt.subplot(1, 2, 2)
stats.probplot(residuals_train, dist="norm", plot=plt)
plt.title("Gráfico Q-Q de Residuos (Train)")
# Ajustar el diseño de la figura
plt.tight_layout()
plt.show()