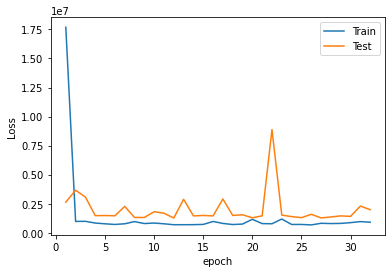
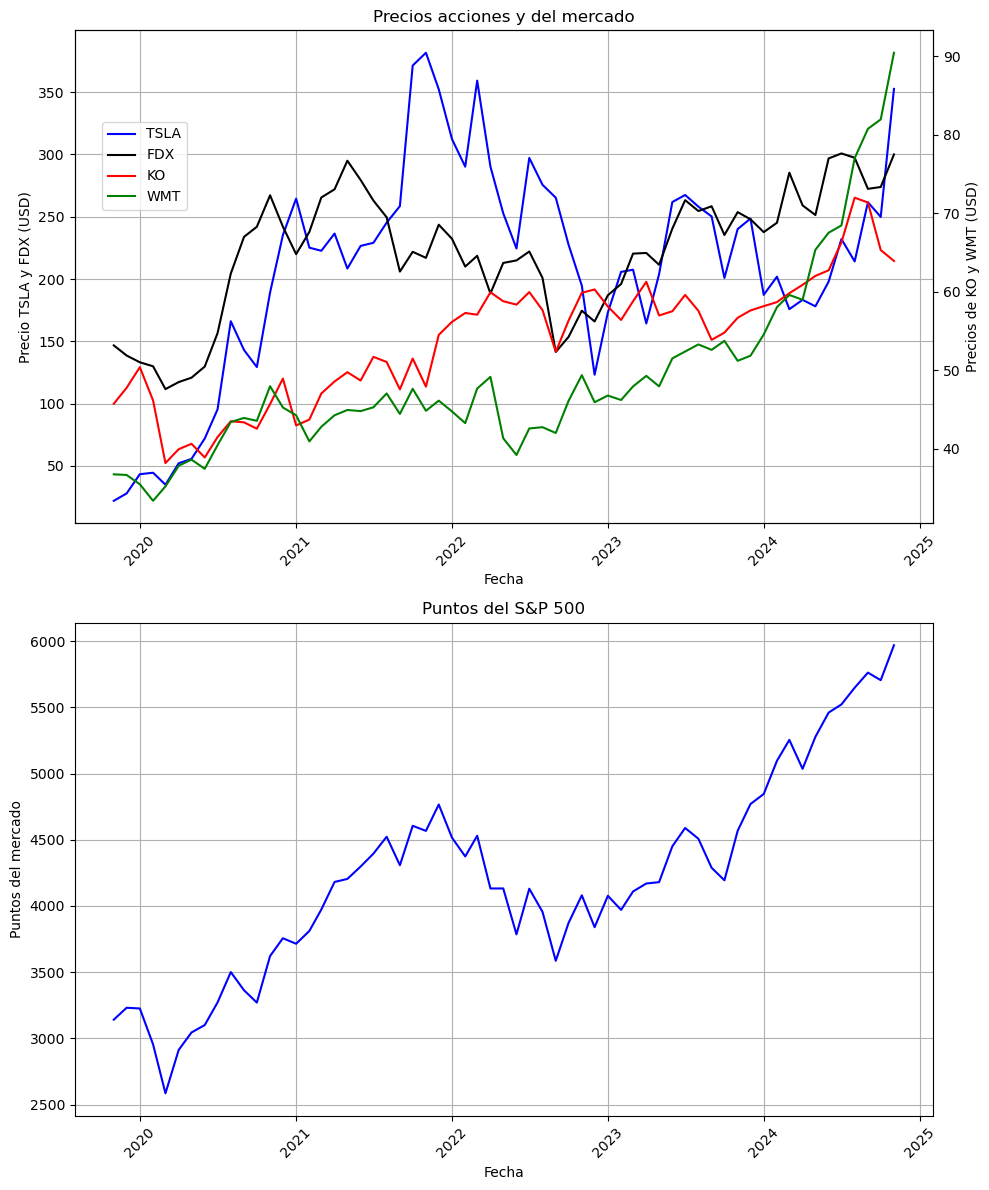
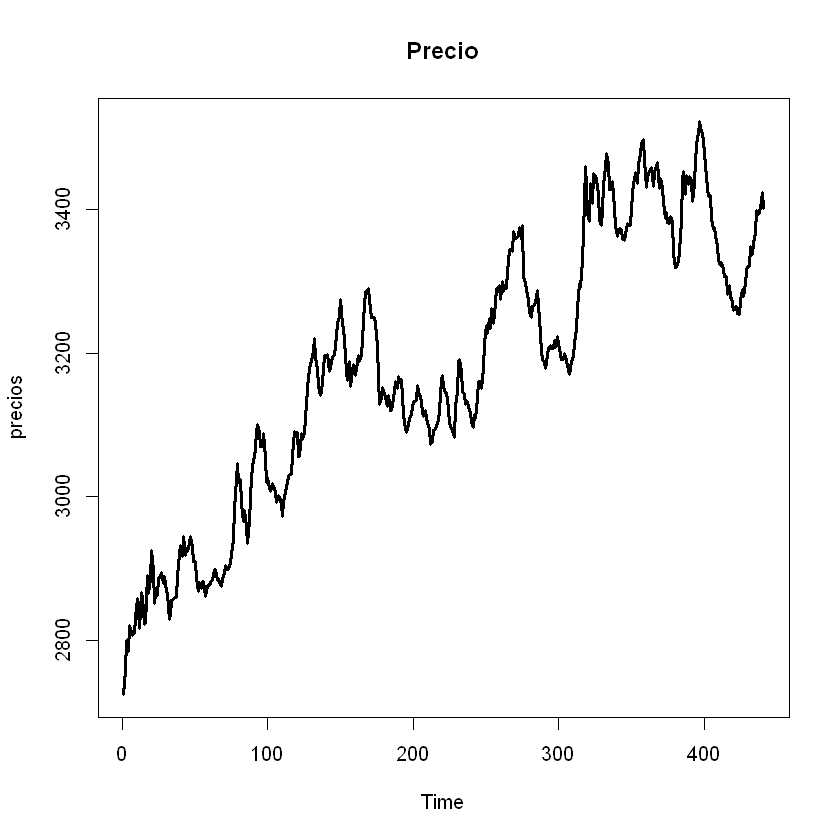
Ejemplo árboles de decisión#
Generación de datos Ficticios:#
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.datasets import make_classification
from sklearn.tree import DecisionTreeClassifier
from sklearn import tree
from sklearn.metrics import accuracy_score, confusion_matrix, classification_report
import seaborn as sns
# Función para agregar ruido y datos atípicos
def add_noise_and_outliers(
X, y, noise_level=0.1, outlier_fraction=0.1, random_state=None
):
np.random.seed(random_state) # Fijar semilla para la reproducibilidad
# Agregar ruido
noise = np.random.normal(0, noise_level, X.shape)
X_noisy = X + noise
# Agregar datos atípicos
n_samples, n_features = X.shape
n_outliers = int(n_samples * outlier_fraction)
outliers = np.random.uniform(low=-4, high=4, size=(n_outliers, n_features))
# Combinar datos originales y atípicos
X_outliers = np.vstack([X_noisy, outliers])
y_outliers = np.hstack([y, np.random.randint(0, 2, size=n_outliers)])
return X_outliers, y_outliers
# Generar datos ficticios con una semilla fija
X, y = make_classification(
n_samples=100,
n_features=2,
n_informative=2,
n_redundant=0,
n_clusters_per_class=1,
random_state=50,
)
# Agregar ruido y datos atípicos con una semilla fija
X_noisy, y_noisy = add_noise_and_outliers(
X, y, noise_level=0.5, outlier_fraction=0.2, random_state=50
)
# Convertir a DataFrame para mejor visualización
df = pd.DataFrame(X_noisy, columns=["Variable 1", "Variable 2"])
df["Label"] = y_noisy
X = df[["Variable 1", "Variable 2"]].values
y = df["Label"].values
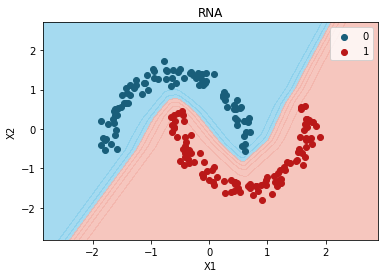
# Visualizar datos con ruido y atípicos
plt.scatter(
df["Variable 1"], df["Variable 2"], c=df["Label"], cmap="viridis", edgecolor="k"
)
plt.xlabel("Variable 1")
plt.ylabel("Variable 2")
plt.title("Datos Ficticios")
plt.show()
Entrenamiento del Árbol:#
Vamos a entrenar un árbol de decisión con pre-poda utilizando los
hiperparámetros max_depth, min_samples_split y
min_samples_leaf.
from sklearn.tree import DecisionTreeClassifier
from sklearn import tree
# Crear modelo con pre-poda
decision_tree = DecisionTreeClassifier(
max_depth=3, min_samples_split=5, min_samples_leaf=2, random_state=34
)
decision_tree.fit(X, y)
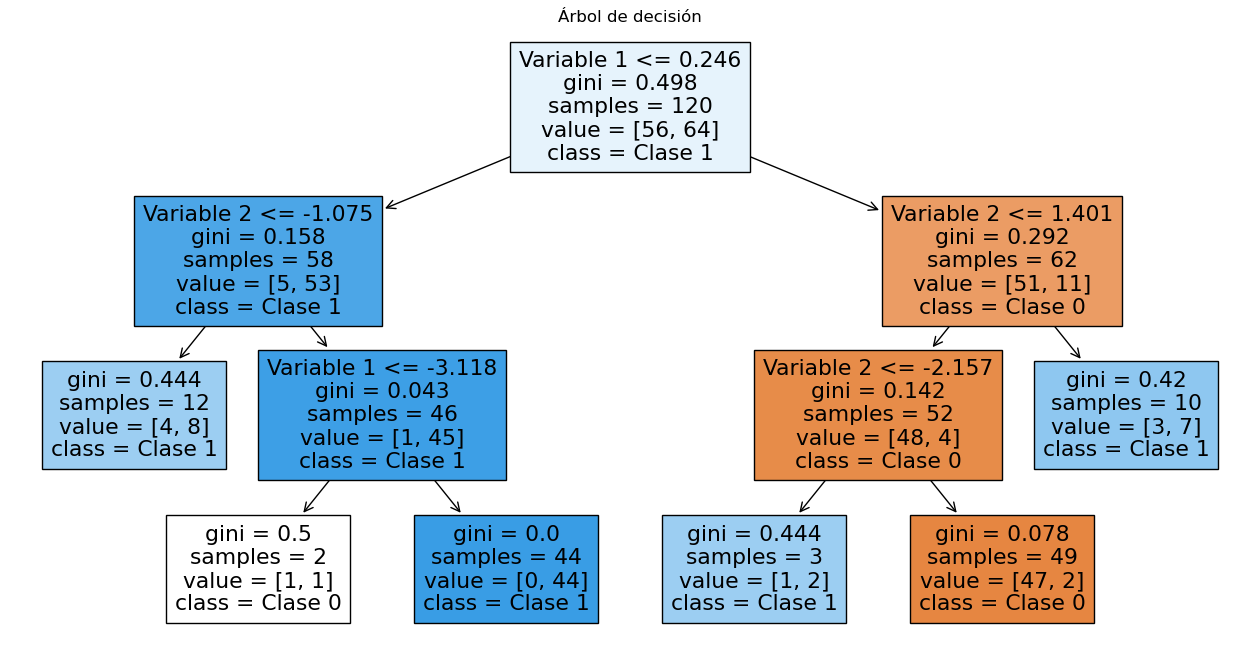
# Visualizar árbol pre-podado
plt.figure(figsize=(16, 8))
tree.plot_tree(
decision_tree,
filled=True,
feature_names=["Variable 1", "Variable 2"],
class_names=["Clase 0", "Clase 1"],
)
plt.title("Árbol de decisión")
plt.show()
Interpretación del índice Gini:
Un índice de Gini de 0,5 indica que el nodo es completamente impuro. En el contexto de un problema de clasificación binaria (con dos clases), un índice de Gini de 0,5 significa que las clases están distribuidas equitativamente en el nodo. Es decir, la probabilidad de escoger un elemento de cualquier clase es igual.
Esto se da cuando:
Hay un 50% de elementos de una clase y un 50% de elementos de la otra clase en el nodo.
En términos más generales, para un nodo con \(n\) clases, un índice de Gini de 0,5 sugiere que la clase de los elementos es completamente incierta, lo que hace que la partición no sea útil para la clasificación.
Con un índice de Gini de 0,498 hay casi un 50% de probabilidad de encontrar cualquiera de las dos clases en ese nodo.
Por otro lado, un índice de Gini de 0,292 indica que el nodo tiene una mayor proporción de elementos de una clase en comparación con la otra, lo que significa que el nodo es relativamente puro. La impureza se reduce porque hay una mayor concentración de elementos de una sola clase, aunque no es completamente puro.
Evaluación del modelo:#
# Predicciones
y_pred = decision_tree.predict(X)
# Evaluación del modelo
accuracy = accuracy_score(y, y_pred)
conf_matrix = confusion_matrix(y, y_pred)
class_report = classification_report(y, y_pred)
print(f"Precisión del modelo: {accuracy}")
print("Informe de clasificación:")
print(class_report)
# Visualizar matriz de confusión
plt.figure(figsize=(8, 6))
sns.heatmap(
conf_matrix,
annot=True,
fmt="d",
cmap="Blues",
xticklabels=["Clase 0", "Clase 1"],
yticklabels=["Clase 0", "Clase 1"],
)
plt.xlabel("Predicted")
plt.ylabel("Actual")
plt.title("Matriz de Confusión")
plt.show()
Precisión del modelo: 0.9416666666666667
Informe de clasificación:
precision recall f1-score support
0 0.92 0.96 0.94 56
1 0.97 0.92 0.94 64
accuracy 0.94 120
macro avg 0.94 0.94 0.94 120
weighted avg 0.94 0.94 0.94 120
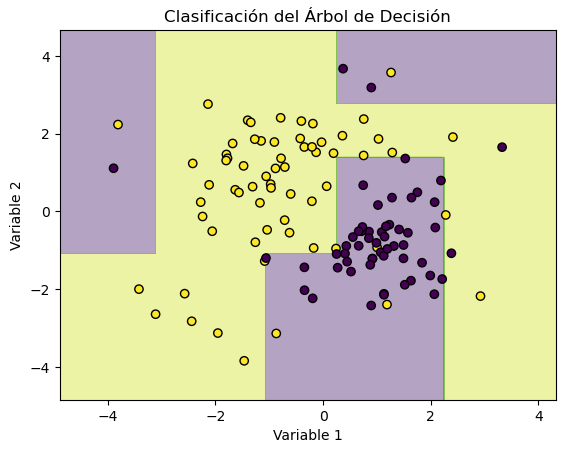
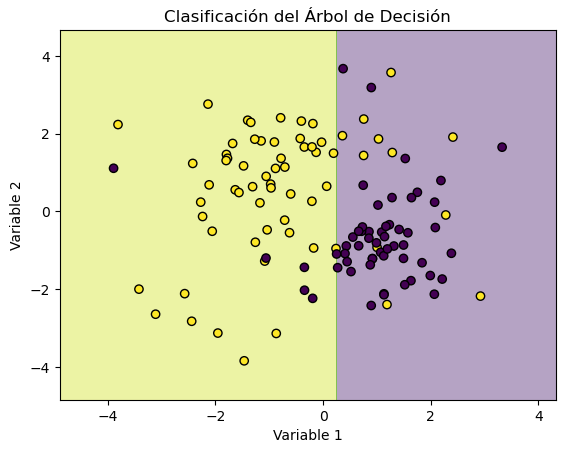
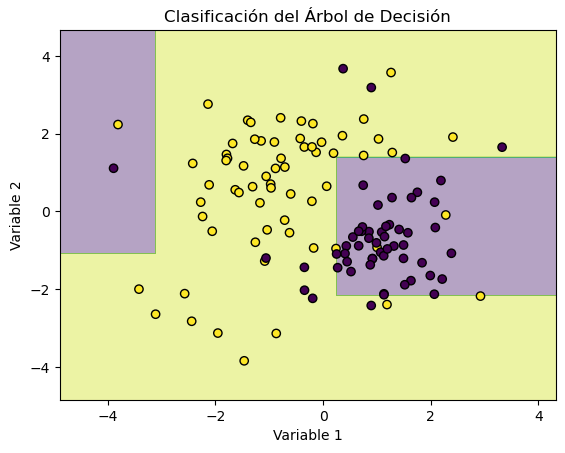
# Crear una malla de puntos para el gráfico
x_min, x_max = X[:, 0].min() - 1, X[:, 0].max() + 1
y_min, y_max = X[:, 1].min() - 1, X[:, 1].max() + 1
xx, yy = np.meshgrid(np.arange(x_min, x_max, 0.01), np.arange(y_min, y_max, 0.01))
# Predecir las clases para cada punto en la malla
Z = decision_tree.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
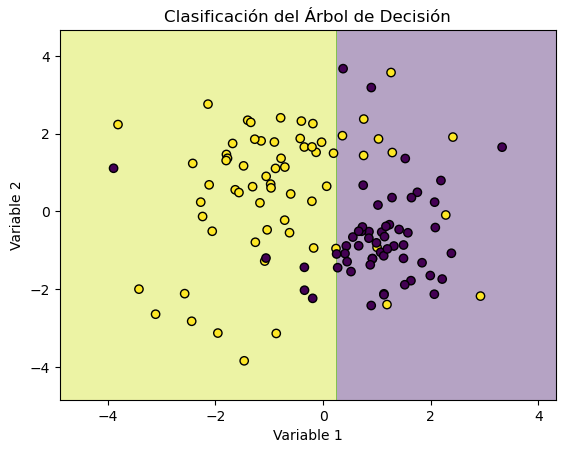
# Visualizar la clasificación
plt.contourf(xx, yy, Z, alpha=0.4, cmap="viridis")
plt.scatter(X[:, 0], X[:, 1], c=y, edgecolors="k", marker="o", cmap="viridis")
plt.xlabel("Variable 1")
plt.ylabel("Variable 2")
plt.title("Clasificación del Árbol de Decisión")
plt.show()
Efecto de cambiar los hiperparámetros:#
max_depth bajo:
# Crear modelo con pre-poda
decision_tree = DecisionTreeClassifier(
max_depth=1, min_samples_split=2, min_samples_leaf=2, random_state=34
)
decision_tree.fit(X, y)
# Predicciones
y_pred = decision_tree.predict(X)
# Evaluación del modelo
accuracy = accuracy_score(y, y_pred)
print(f"Precisión del modelo: {accuracy}")
# Visualizar árbol pre-podado
plt.figure(figsize=(16, 8))
tree.plot_tree(
decision_tree,
filled=True,
feature_names=["Variable 1", "Variable 2"],
class_names=["Clase 0", "Clase 1"],
)
plt.title("Árbol de decisión")
plt.show()
# Crear una malla de puntos para el gráfico
x_min, x_max = X[:, 0].min() - 1, X[:, 0].max() + 1
y_min, y_max = X[:, 1].min() - 1, X[:, 1].max() + 1
xx, yy = np.meshgrid(np.arange(x_min, x_max, 0.01), np.arange(y_min, y_max, 0.01))
# Predecir las clases para cada punto en la malla
Z = decision_tree.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
# Visualizar la clasificación
plt.contourf(xx, yy, Z, alpha=0.4, cmap="viridis")
plt.scatter(X[:, 0], X[:, 1], c=y, edgecolors="k", marker="o", cmap="viridis")
plt.xlabel("Variable 1")
plt.ylabel("Variable 2")
plt.title("Clasificación del Árbol de Decisión")
plt.show()
Precisión del modelo: 0.8666666666666667

max_depth alto:
# Crear modelo con pre-poda
decision_tree = DecisionTreeClassifier(
max_depth=5, min_samples_split=2, min_samples_leaf=2, random_state=34
)
decision_tree.fit(X, y)
# Predicciones
y_pred = decision_tree.predict(X)
# Evaluación del modelo
accuracy = accuracy_score(y, y_pred)
print(f"Precisión del modelo: {accuracy}")
# Visualizar árbol pre-podado
plt.figure(figsize=(16, 8))
tree.plot_tree(
decision_tree,
filled=True,
feature_names=["Variable 1", "Variable 2"],
class_names=["Clase 0", "Clase 1"],
)
plt.title("Árbol de decisión")
plt.show()
# Crear una malla de puntos para el gráfico
x_min, x_max = X[:, 0].min() - 1, X[:, 0].max() + 1
y_min, y_max = X[:, 1].min() - 1, X[:, 1].max() + 1
xx, yy = np.meshgrid(np.arange(x_min, x_max, 0.01), np.arange(y_min, y_max, 0.01))
# Predecir las clases para cada punto en la malla
Z = decision_tree.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
# Visualizar la clasificación
plt.contourf(xx, yy, Z, alpha=0.4, cmap="viridis")
plt.scatter(X[:, 0], X[:, 1], c=y, edgecolors="k", marker="o", cmap="viridis")
plt.xlabel("Variable 1")
plt.ylabel("Variable 2")
plt.title("Clasificación del Árbol de Decisión")
plt.show()
Precisión del modelo: 0.9416666666666667
min_samples_split alto:
# Crear modelo con pre-poda
decision_tree = DecisionTreeClassifier(
max_depth=3, min_samples_split=20, min_samples_leaf=2, random_state=34
)
decision_tree.fit(X, y)
# Predicciones
y_pred = decision_tree.predict(X)
# Evaluación del modelo
accuracy = accuracy_score(y, y_pred)
print(f"Precisión del modelo: {accuracy}")
# Visualizar árbol pre-podado
plt.figure(figsize=(16, 8))
tree.plot_tree(
decision_tree,
filled=True,
feature_names=["Variable 1", "Variable 2"],
class_names=["Clase 0", "Clase 1"],
)
plt.title("Árbol de decisión")
plt.show()
# Crear una malla de puntos para el gráfico
x_min, x_max = X[:, 0].min() - 1, X[:, 0].max() + 1
y_min, y_max = X[:, 1].min() - 1, X[:, 1].max() + 1
xx, yy = np.meshgrid(np.arange(x_min, x_max, 0.01), np.arange(y_min, y_max, 0.01))
# Predecir las clases para cada punto en la malla
Z = decision_tree.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
# Visualizar la clasificación
plt.contourf(xx, yy, Z, alpha=0.4, cmap="viridis")
plt.scatter(X[:, 0], X[:, 1], c=y, edgecolors="k", marker="o", cmap="viridis")
plt.xlabel("Variable 1")
plt.ylabel("Variable 2")
plt.title("Clasificación del Árbol de Decisión")
plt.show()
Precisión del modelo: 0.9083333333333333
min_samples_leaf alto:
# Crear modelo con pre-poda
decision_tree = DecisionTreeClassifier(
max_depth=3, min_samples_split=5, min_samples_leaf=10, random_state=34
)
decision_tree.fit(X, y)
# Predicciones
y_pred = decision_tree.predict(X)
# Evaluación del modelo
accuracy = accuracy_score(y, y_pred)
print(f"Precisión del modelo: {accuracy}")
# Visualizar árbol pre-podado
plt.figure(figsize=(16, 8))
tree.plot_tree(
decision_tree,
filled=True,
feature_names=["Variable 1", "Variable 2"],
class_names=["Clase 0", "Clase 1"],
)
plt.title("Árbol de decisión")
plt.show()
# Crear una malla de puntos para el gráfico
x_min, x_max = X[:, 0].min() - 1, X[:, 0].max() + 1
y_min, y_max = X[:, 1].min() - 1, X[:, 1].max() + 1
xx, yy = np.meshgrid(np.arange(x_min, x_max, 0.01), np.arange(y_min, y_max, 0.01))
# Predecir las clases para cada punto en la malla
Z = decision_tree.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
# Visualizar la clasificación
plt.contourf(xx, yy, Z, alpha=0.4, cmap="viridis")
plt.scatter(X[:, 0], X[:, 1], c=y, edgecolors="k", marker="o", cmap="viridis")
plt.xlabel("Variable 1")
plt.ylabel("Variable 2")
plt.title("Clasificación del Árbol de Decisión")
plt.show()
Precisión del modelo: 0.9
max_leaf_nodes bajo:
# Crear modelo con pre-poda
decision_tree = DecisionTreeClassifier(
max_depth=3,
min_samples_split=5,
min_samples_leaf=2,
max_leaf_nodes=2,
random_state=34,
)
decision_tree.fit(X, y)
# Predicciones
y_pred = decision_tree.predict(X)
# Evaluación del modelo
accuracy = accuracy_score(y, y_pred)
print(f"Precisión del modelo: {accuracy}")
# Visualizar árbol pre-podado
plt.figure(figsize=(16, 8))
tree.plot_tree(
decision_tree,
filled=True,
feature_names=["Variable 1", "Variable 2"],
class_names=["Clase 0", "Clase 1"],
)
plt.title("Árbol de decisión")
plt.show()
# Crear una malla de puntos para el gráfico
x_min, x_max = X[:, 0].min() - 1, X[:, 0].max() + 1
y_min, y_max = X[:, 1].min() - 1, X[:, 1].max() + 1
xx, yy = np.meshgrid(np.arange(x_min, x_max, 0.01), np.arange(y_min, y_max, 0.01))
# Predecir las clases para cada punto en la malla
Z = decision_tree.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
# Visualizar la clasificación
plt.contourf(xx, yy, Z, alpha=0.4, cmap="viridis")
plt.scatter(X[:, 0], X[:, 1], c=y, edgecolors="k", marker="o", cmap="viridis")
plt.xlabel("Variable 1")
plt.ylabel("Variable 2")
plt.title("Clasificación del Árbol de Decisión")
plt.show()
Precisión del modelo: 0.8666666666666667

max_leaf_nodes alto:
# Crear modelo con pre-poda
decision_tree = DecisionTreeClassifier(
max_depth=3,
min_samples_split=5,
min_samples_leaf=2,
max_leaf_nodes=10,
random_state=34,
)
decision_tree.fit(X, y)
# Predicciones
y_pred = decision_tree.predict(X)
# Evaluación del modelo
accuracy = accuracy_score(y, y_pred)
print(f"Precisión del modelo: {accuracy}")
# Visualizar árbol pre-podado
plt.figure(figsize=(16, 8))
tree.plot_tree(
decision_tree,
filled=True,
feature_names=["Variable 1", "Variable 2"],
class_names=["Clase 0", "Clase 1"],
)
plt.title("Árbol de decisión")
plt.show()
# Crear una malla de puntos para el gráfico
x_min, x_max = X[:, 0].min() - 1, X[:, 0].max() + 1
y_min, y_max = X[:, 1].min() - 1, X[:, 1].max() + 1
xx, yy = np.meshgrid(np.arange(x_min, x_max, 0.01), np.arange(y_min, y_max, 0.01))
# Predecir las clases para cada punto en la malla
Z = decision_tree.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
# Visualizar la clasificación
plt.contourf(xx, yy, Z, alpha=0.4, cmap="viridis")
plt.scatter(X[:, 0], X[:, 1], c=y, edgecolors="k", marker="o", cmap="viridis")
plt.xlabel("Variable 1")
plt.ylabel("Variable 2")
plt.title("Clasificación del Árbol de Decisión")
plt.show()
Precisión del modelo: 0.9416666666666667