Ejemplo clustering jerárquico#
Importar librerías:#
import numpy as np
import matplotlib.pyplot as plt
from scipy.cluster.hierarchy import dendrogram, linkage, fcluster, cophenet
from sklearn.cluster import AgglomerativeClustering
from scipy.spatial.distance import pdist
Generar datos en forma de espiral:#
def generate_spiral_data(n_points, noise=0.5):
theta = np.sqrt(np.random.rand(n_points)) * 2 * np.pi # Random theta
r_a = 2 * theta + np.pi # Radius for spiral A
data_a = np.array([np.cos(theta) * r_a, np.sin(theta) * r_a]).T
data_a += np.random.randn(n_points, 2) * noise
r_b = -2 * theta - np.pi # Radius for spiral B
data_b = np.array([np.cos(theta) * r_b, np.sin(theta) * r_b]).T
data_b += np.random.randn(n_points, 2) * noise
res_a = np.append(data_a, np.zeros((n_points, 1)), axis=1)
res_b = np.append(data_b, np.ones((n_points, 1)), axis=1)
res = np.append(res_a, res_b, axis=0)
np.random.shuffle(res)
return res[:, :2], res[:, 2]
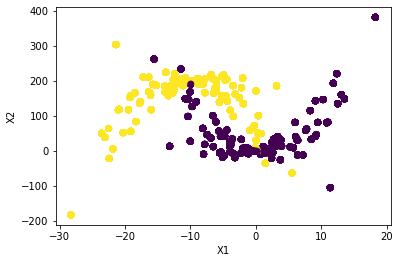
# Generar datos
X, y = generate_spiral_data(1000)
# Visualizar los datos y los clusters
plt.figure(figsize=(10, 7))
plt.scatter(X[:, 0], X[:, 1], cmap="rainbow", alpha=0.7, edgecolors="b")
plt.title("Espiral")
plt.xlabel("X1")
plt.ylabel("X2")
plt.show()
Aplicar clustering jerárquico:#
Usamos AgglomerativeClustering de sklearn.cluster para realizar
clustering aglomerativo; sin embargo, no genera dendrogramas
directamente; se enfoca en la asignación de clusters.
Usamos linkage de scipy.cluster.hierarchy para la matriz de
enlaces y luego visualizar el dendograma.
# Aplicar clustering jerárquico usando el método Ward
model = AgglomerativeClustering(n_clusters=None, distance_threshold=0, linkage="ward")
model = model.fit(X)
# Crear la matriz de enlaces usando el método Ward
Z = linkage(X, method="ward")
Usar n_clusters=None en AgglomerativeClustering indica que no
estamos especificando un número fijo de clusters a priori. En su lugar,
estamos utilizando distance_threshold=0, lo que significa que
queremos construir el dendrograma completo sin realizar un corte
inicial. Esto es útil cuando queremos analizar la estructura jerárquica
completa de los datos antes de decidir cuántos clusters utilizar.
Visualizar el dendrograma:#
# Plot del dendrograma
plt.figure(figsize=(10, 7))
plt.title("Dendrograma")
dendrogram(Z)
plt.show()
Verificación del árbol:#
# Calcular distancias originales
distances = pdist(X)
# Calcular la correlación coefénica
c, coph_dists = cophenet(Z, distances)
print(f"Correlación cofenética: {c:.4f}")
Correlación cofenética: 0.7240
Calcular distancias coefénicas: Utilizamos pdist para calcular
las distancias originales entre los puntos y cophenet para calcular
la correlación entre las distancias cofenéticas y las distancias
originales.
Una correlación cercana a 1 indica una buena representación.
Una vez que tenemos el dendrograma completo, podemos decidir dónde cortar el dendrograma para obtener el número deseado de clusters. Esto se puede hacer visualmente o utilizando criterios específicos basados en la distancia.
Asignación cantidad de clusters:#
# Asignar clusters
n_clusters = 2 # Definir el número de clusters
model = AgglomerativeClustering(n_clusters=n_clusters, linkage="ward")
labels = model.fit_predict(X)
# Visualizar los datos y los clusters
plt.figure(figsize=(10, 7))
plt.scatter(X[:, 0], X[:, 1], c=labels, cmap="rainbow", alpha=0.7, edgecolors="b")
plt.title("Clustering Jerárquico")
plt.xlabel("X1")
plt.ylabel("X2")
plt.show()
Determinación del número de clusters usando un umbral de distancia:#
# Establecer un umbral de distancia
distance_threshold = 200 # Umbral basado en la distancia donde cortar el dendrograma
# Determinar los clusters usando el umbral de distancia
clusters = fcluster(Z, t=distance_threshold, criterion="distance")
# Visualizar los datos y los clusters
plt.figure(figsize=(10, 7))
plt.scatter(X[:, 0], X[:, 1], c=clusters, cmap="rainbow", alpha=0.7, edgecolors="b")
plt.title(f"Clustering Jerárquico con Umbral de Distancia {distance_threshold}")
plt.xlabel("X1")
plt.ylabel("X2")
plt.show()
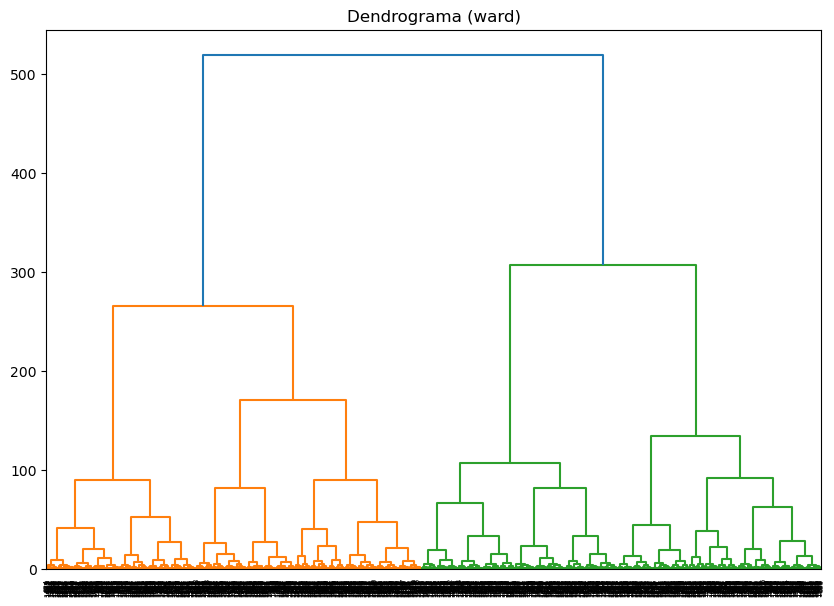
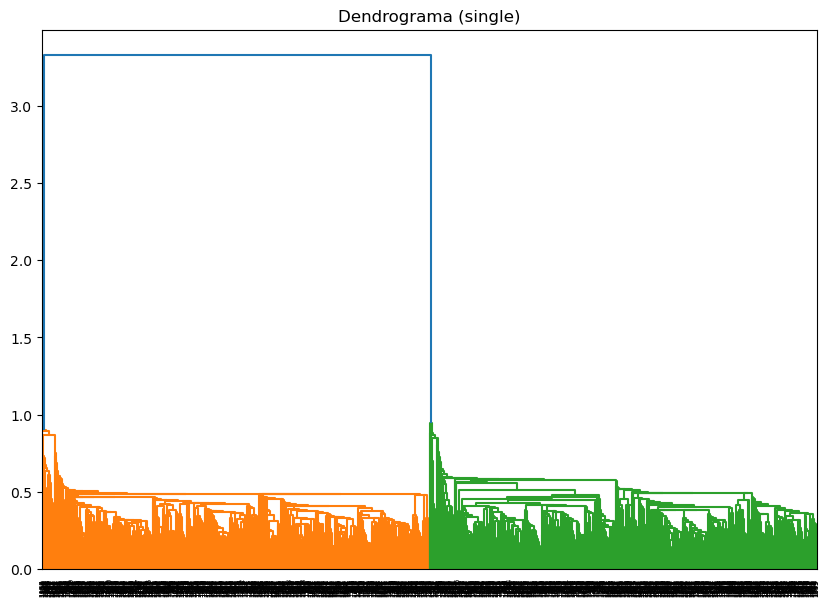
Implementación de métodos alternativos de vinculación:#
# Métodos de vinculación a probar
linkage_methods = ["single", "complete", "average", "ward"]
for method in linkage_methods:
# Crear la matriz de enlaces
Z = linkage(X, method=method)
# Calcular la correlación coefénica
c, coph_dists = cophenet(Z, pdist(X))
print(f"Método de vinculación: {method}, Correlación cofenética: {c:.4f}")
# Plot del dendrograma
plt.figure(figsize=(10, 7))
plt.title(f"Dendrograma ({method})")
dendrogram(Z)
plt.show()
Método de vinculación: single, Correlación cofenética: 0.2346
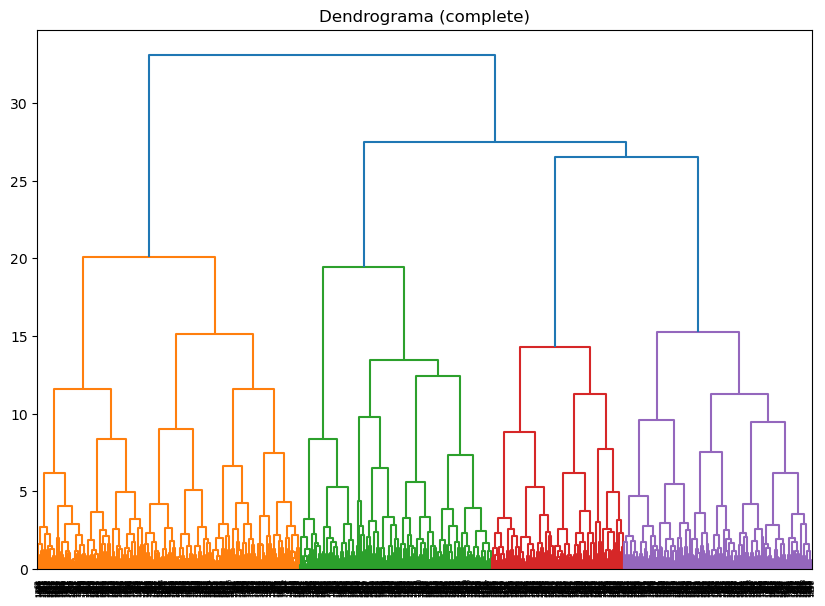
Método de vinculación: complete, Correlación cofenética: 0.7171
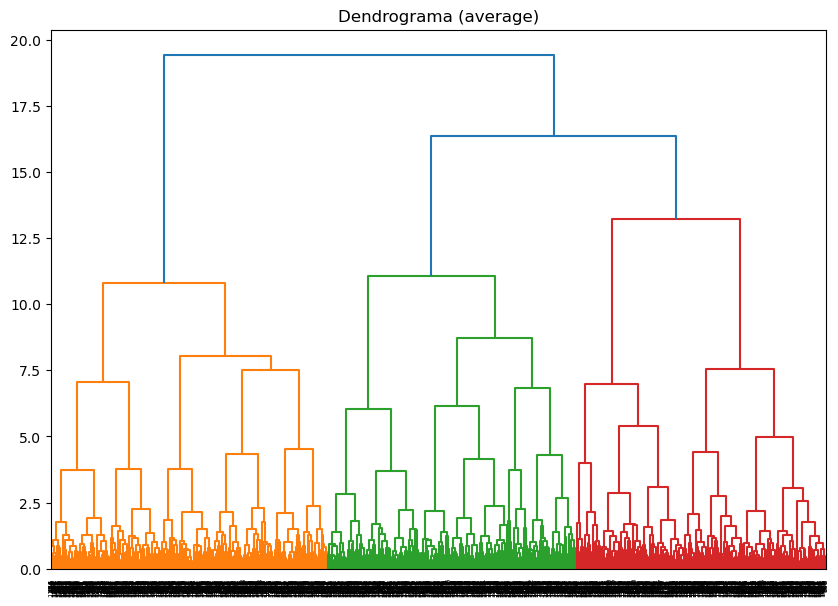
Método de vinculación: average, Correlación cofenética: 0.7219
Método de vinculación: ward, Correlación cofenética: 0.7240